Méthodes pour l’étude de structures, dynamiques et interactions d’acides nucléiques non canoniques
Introduction
“In France, everything has an origin.” Chercheur canadien, 11/2025
Depuis mes premiers travaux de doctorat, jusqu’à mes travaux les plus récents, j’ai eu la chance de travailler dans des environnements très divers : recherche fondamentale en milieu académique, ou appliquée dans le milieu très régulé de l’industrie pharmaceutique, en France comme à l’étranger, dans des équipes de chimie organique, physique et analytique, de biochimie et de biophysique. À chaque étape, j’ai eu l’opportunité de m’essayer à une large gamme de techniques : synthèse organique et inorganique, développement en sciences analytiques (HPLC, MS, fluorescence), chémoinformatique, modélisation moléculaire, ou encore développement informatique. Sous cette diversité méthodologique se cache un fil conducteur fort : celui de comprendre les forces qui régissent les structures et les interactions non covalentes de macromolécules biologiques, en particulier de l’ADN, et les exploiter à des fins thérapeutiques. Certains de mes travaux les plus récents à l’Université de Bordeaux se penchent sur des questions que j’avais abordées il y a maintenant dix-sept ans, mais de nouvelles questions sont aussi, bien sûr, au centre de mon attention.
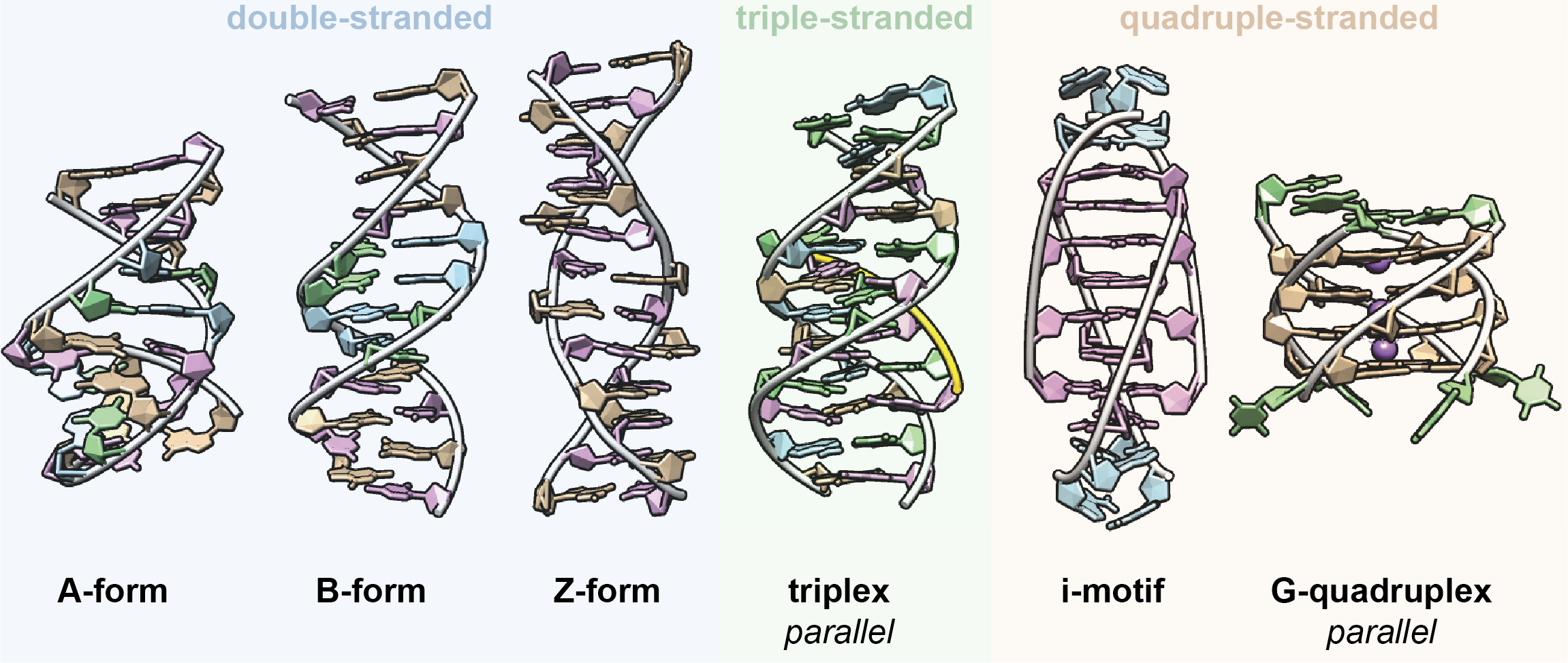
Dès mes premiers travaux de thèse, j’ai développé un amour certain pour les structures non canoniques de l’ADN (Figure 1). Celles qui ne forment pas de doubles hélices, celles qui se moquent de Watson et Crick (et Franklin), celles qui ont des bases et des sucres bizarres. Et, soyons honnêtes, tout particulièrement les G-quadruplexes (G4s). Tous les travaux que je décris ci-après ne concernent pas forcément ces bizarreries, il est même question de protéines (!), mais le lecteur attentif remarquera que ces écarts ont servi de sources d’inspiration a mes amours originelles. Reculer pour mieux sauter en somme.
Parmi les autres passions se sont révélées à moi et serviront également de fil rouge dans ce manuscrit, deux sont notables :
L’automatisation du travail. J’ai rapidement fait le constat que la fainéantise, plutôt que la nécessité, est mère de l’invention, et qu’en automatisant et/ou accélérant les processus d’analyse je pourrais me consacrer à d’autres entreprises.1 Plus sérieusement, j’y ai trouvé de nombreux avantages : efficacité, reproductibilité, capacité à gérer des sets de données très larges et à générer des résultats (figures, rapports) standardisés. Mon expérience en milieu industriel régulé n’y est pas non plus étranger.
Faire de la science ouverte et accessible : produire des figures claires et sans biais, rendre les données brutes et les méthodes/code de leur analyse publique.
1 Pour entrevoir l’essentiel, il ne faut exercer aucun métier, nous dit Emil Cioran.
Mes diverses expériences m’ont permis de rencontrer et travailler avec des personnes formidables. Si elles m’ont fréquemment laissé l’impression d’être intellectuellement limité, elles m’ont surtout beaucoup appris, tant scientifiquement qu’humainement. En écrivant ce manuscrit, j’ai eu envie de visualiser mon réseau de co-auteurs (Figure 2). Les clusters qui se forment racontent finalement assez bien ma carrière (que je survole ; ci-après). J’ai aussi découvert que je n’avais jamais rencontré certains d’entre eux et qu’en recherche comme dans la vie, les amis de mes amis sont mes amis. Quoi qu’il en soit, je les remercie tous.
Enfin, j’aimerais remercier ici tous les étudiants que j’ai eu la chance d’encadrer. Je suis particulièrement heureux que mes derniers stagiaires de master/école d’ingénieur se soient lancés dans des thèses, malgré mon insistance à leur dire que ce n’est pas un bon choix de carrière. Je ne suis sans doute pas pour grand chose dans leur réussite mais je ne les aurais au moins pas dégouttés de la recherche.
Résumé du déroulé de carrière
Mes études supérieures ont débutées par l’obtention d’un diplôme d’ingénieur chimiste à l’École Supérieure de Chimie Organique et Minérale, (à l’époque) à Cergy-Pontoise (Figure 3). J’ai complété ma formation initiale par un Master 2 recherche Chimie fine, analytique et polymères de l’Université de Cergy-Pontoise, effectué en parallèle de ma dernière année d’école d’ingénieur.
Mon stage de M2, qui a également fait office de stage de fin d’étude d’ingénieur, sur le développement d’une méthode de synthèse de di-organozinciques symétriques, est en apparence peu en rapport avec ma carrière académique et n’est pas détaillé ci-après. Les apparences peuvent être trompeuses, car ce sont partiellement ces travaux qui m’ont amené vers mon sujet de thèse, initialement centré sur la synthèse de complexes de métaux de transition comme ligand d’ADN G-quadruplex. L’autre étape de mes études m’ayant conduit à postuler à ce sujet de thèse est un stage estival chez Sanofi-Aventis (équipe de chimie exploratoire ; site de Vitry-sur-Seine), en 2007, pendant lequel j’ai découvert le travail de chimiste à l’interface avec la biologie - aspect qui était absent de ma formation académique jusque-là. J’y ai préparé des conjugués entre le fragment FAB de l’Herceptin et des fluorophores ou molécules cages (pour la coordination d’isotopes radioactif d’indium) à destination de l’imagerie médicale de petits mammifères. Ce stage présagera d’autres de mes activités futures : caractérisation d’anticorps par chromatographie d’exclusion stérique, spectrométrie de masse, et conjugaison de fluorophores.
J’ai obtenu mon doctorat de chimie de l’Université Paris XI en novembre 2011 grâce à mes travaux de thèse à l’Institut Curie, sous la direction du Dr. Marie-Paule Teulade-Fichou, à l’époque directrice de l’UMR 176 du CNRS. Pour cette thèse, Développement de méthodes analytiques pour le criblage haut débit de ligands d’ADN. Synthèse de petites molécules ligands d’ADN G quadruplex à visée anticancéreuse, j’ai obtenu une bourse de docteur ingénieur (BDI) co-financée par le CNRS et l’Institut Curie (1 Doctorat à l’Institut Curie/CNRS UMR176/Université Paris XI). Je suis ensuite resté dans l’équipe jusqu’en juillet 2012 comme chercheur postdoctorant, ce qui m’a permis de terminer plusieurs projets entrepris pendant ma thèse. Pendant ces presque quatre ans, j’ai complété ma mue de la chimie organique vers la chimie physique et analytique.
J’ai ensuite rejoins le laboratoire du Dr. David Perrin, du département de chimie de l’Université de Colombie Britannique (UBC), au Canada (2 Postdoctorat à l’Université de Colombie Britannique). Dans cette équipe, dont les intérêts sont partagés entre synthèse organique et la sélection d’aptamères d’ADN fonctionnalisés, par SELEX, j’ai continué mon apprentissage des méthodes biophysiques pour caractériser des structures d’acides nucléiques, et étendu mon répertoire méthodologique aux calculs de DFT et dynamiques moléculaires. J’ai financé ce séjour postdoctoral grâce à une bourse Banting (140 k$).
Après une année à UBC sevré de G4s, je suis revenu en France, plus précisément à Bordeaux, dans les équipes de Jean-Louis Mergny et Valérie Gabelica à l’Institut Européen de Chimie et Biologie (IECB, ARNA, INSERM U869 ; Figure 4). Je me suis à nouveau intéressé aux structures non canoniques de l’ADN, à l’interface entre ces deux équipes (3 Postdoctorat à l’Institut Européen de Chimie et Biologie). Dans un premier temps, j’ai entrepris avec Jean-Louis le développement d’une méthode de SEC-HPLC pour caractériser des structures d’oligonucléotides structurés ; idée du regretté Jean-Louis Leroy (ICSN, Gif-sur-Yvette). Je me suis ensuite penché sur le rôle des cations dans la structuration des G4s. Ce sujet d’étude m’a permis de passer de l’équipe de Jean-Louis à celle de Valérie, et d’y découvrir la spectrométrie de masse native (après une courte mais intense initiation en thèse).
J’ai ensuite quitté le monde académique avant même d’avoir tenté ma chance aux concours. J’ai en effet accepté un poste de chercheur dans l’industrie pharmaceutique, dans laquelle j’ai mené à bien un projet financé par l’Union Européenne (Fonds européen de développement régional, « FEDER »). Cela m’a permis de mettre en œuvre un large éventail de méthodes de caractérisation et de quantification des protéines et des petites molécules thérapeutiques dans un environnement soumis à des réglementations très strictes, et avec des objectifs, des moyens et une temporalité différents de ceux de la recherche académique (4 R&D Scientist à Quality Assistance).
Mes travaux industriels, bien que n’impliquant pas d’acides nucléiques, ont été une source d’inspiration pour l’analyse de ces derniers, en particulier l’utilisation de l’HDX/MS pour la caractérisation de structures d’oligonucléotides et de leurs interactions avec des protéines et petites molécules. Cette expérience constitue également une base importante de mes enseignements, ainsi que de la formation continue que j’offre aux étudiants que je supervise au laboratoire (bonnes pratiques de fabrication, connaissance du monde industriel).
L’ensemble de ces travaux de recherche antérieurs à ma prise de poste à l’université de Bordeaux m’ont progressivement mené de la synthèse de petites molécules pour le ciblage de bio-macromolécules d’intérêt thérapeutique, vers la caractérisation physico-chimique et computationnelle de ces bio-macromolécules et de leurs interactions, formant un ensemble cohérent, complémentaire et en adéquation avec les projets de recherche que je développe actuellement.
Dernière étape : je suis depuis 2017 maître de conférences à l’Université de Bordeaux, rattaché à l’UFR de pharmacie pour l’enseignement et à l’unité ARNA (INSERM U1212, CNRS UMR5320) pour la recherche (Figures 5, 6). Je suis ainsi revenu à l’IECB, dans l’équipe de recherche de V. Gabelica, BALI (Biophysics Analysis and Ligands), mais avec de nouvelles thématiques (5 Thèmes de recherche à l’Université de Bordeaux : résultats et perspectives). Suite au départ de Valérie pour l’Université de Genève, j’ai rejoint l’équipe PRISM de Cameron Mackereth fin 2023. J’y poursuis le travail engagé dans l’équipe de Valérie mais en explore aussi de nouvelles.
Ce mémoire est partiellement chronologique. Si les grands chapitres suivent le fil de ma carrière, j’ai regroupé les travaux par thématiques au sein de ces chapitres pour en faciliter la lecture.