3 Postdoctorat à l’Institut Européen de Chimie et Biologie
A mon retour du Canada, j’ai travaillé comme post-doctorant INSERM à l’Institut Européen de Chimie et de Biologie dans les équipes de J.-L. Mergny et V. Gabelica (Figure 3.1). Je me suis à nouveau intéressé aux structures non canoniques de l’ADN, en développant et utilisant des techniques de chromatographie, spectrométrie de masse native et spectroscopie, à l’interface entre ces deux équipes. Je présente ci-dessous les deux principaux projets que j’ai menés.
3.1 Chromatographie d’exclusion stérique pour l’étude du polymorphisme des acides nucléiques
De nombreux laboratoires académiques s’intéressent aux structures non-canoniques des acides nucléiques et à leurs rôles dans le vivant, comme en témoignent les précédentes sections. Dans le même temps, l’industrie pharmaceutique développe de plus en plus d’acides nucléiques thérapeutiques et a un donc des besoins grandissants de caractérisation de leurs structures secondaires [1–4].
À mon arrivée dans son équipe, Jean-Louis Mergny m’a proposé de développer une méthode de chromatographie d’exclusion stérique (SEC-HPLC) pour discriminer des oligonucléotides en fonction de leur structure secondaire (ssDNA, dsDNA, i-motif, triplex, quadruplex) ainsi que de quantifier leur potentiel autoassemblage en multimères [5].
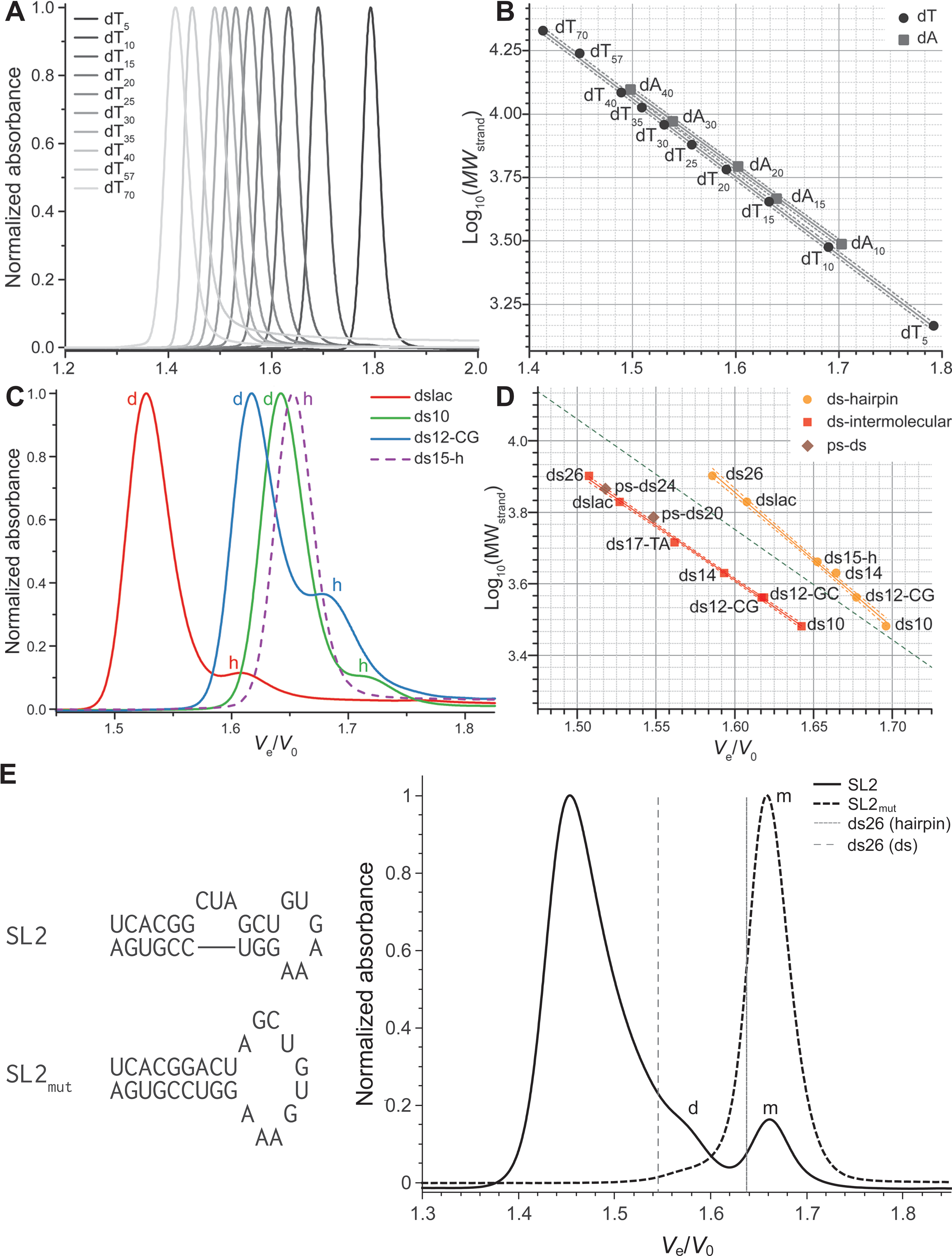
Je me suis appuyé sur une calibration externe extensive, impliquant plus de 110 oligonucléotides formant de nombreuses structures secondaires (duplexes antiparallèles, duplexes parallèles, duplexes A-tract courbés, duplexes mésappariés, triplexes, G4s, i-motifs, tiges-boucles d’ARN). En particulier, les oligonucléotides non structurés ou structurés en duplexes donnent des courbes de calibration possédant d’excellentes linéarités, ce qui permet de caractériser leur longueur et stœchiométrie avec une très grande confiance (Figure 3.2).
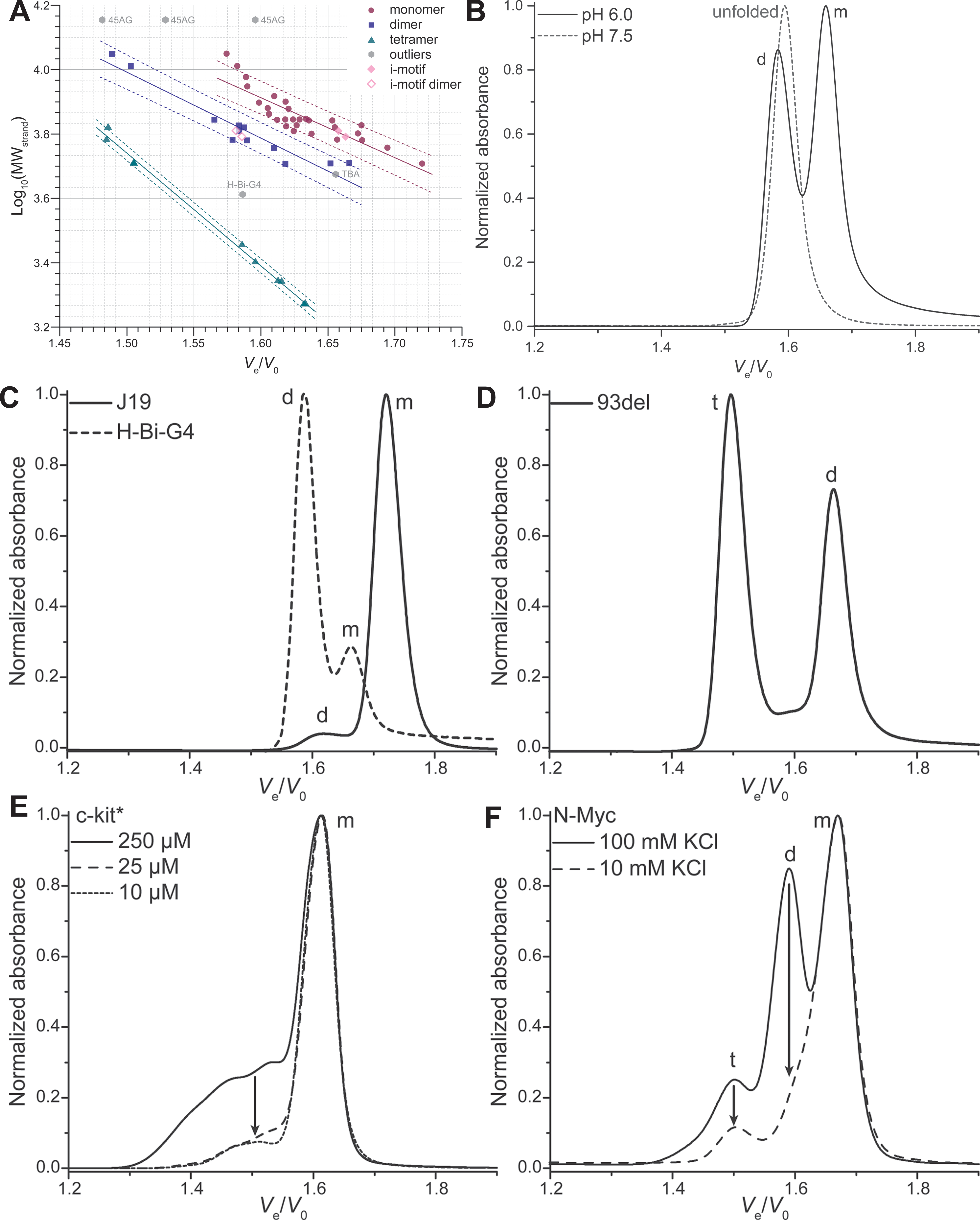
Comme je l’ai déjà évoqué dans le Chapitre 1, les G4s sont particulièrement polymorphes. La plus faible linéarité de la calibration n’a donc pas été une très grande surprise (Figure 3.3A). Cela dit, nous avons montré que la stœchiométrie des G4s pouvait être déterminée avec un haut niveau de confiance, tout comme leur structuration (Figure 3.3B-D).
Par ailleurs, et c’est sans doute l’aspect que je trouve le plus intéressant dans cette technique, la SEC-HPLC permet de rapidement estimer l’influence sur la structure d’acides nucléiques des conditions expérimentales (concentrations en oligonucléotide et sels, nature du tampon et des sels, température, pH), sur une grande gamme dynamique (Figure 3.3B, E, F). Cela est plus difficile, voire impossible, par d’autres méthodes de par l’incompatibilité ce certaines conditions expérimentales : tampons et sels non-volatiles en spectrométrie de masse, trop faibles concentrations en oligonucléotides en RMN, coexistence de plusieurs conformères en cristallographie. La SEC-HPLC est d’ailleurs, je pense, une méthode de choix pour des études multi-techniques pour faire le lien entre des résultats obtenus dans des conditions très différentes ; par exemple des concentrations élevées en oligonucléotides et sels en RMN contre des concentrations plus faibles en spectrométrie de masse (Figure 3.3E, F).
J’ai appliqué cette méthode d’analyse dans d’autres projets, comme dans l’étude de l’influence des riboses sur la stabilité des G4s, où nous avons démontré que les résidus d’ARN ne stabilisent pas forcément ces structures, contrairement à ce qui était supposé jusque-là [12].
Une dernière chose me parait intéressante à propos de cette étude. L’éditeur en charge de cette publication était initialement réticent à l’envoyer en relecture par des pairs et il a fallu faire preuve de conviction pour forcer le destin. Malgré cela, cette publication est fréquemment citée et, surtout, j’ai été plusieurs fois contacté par des industriels pour sa mise en place dans l’industrie pharmaceutique.
3.2 Influence des cations sur les G4s : découverte et caractérisation de doubles commutateurs
Nous l’avons vu, les structures G4 présentent un polymorphisme structurel important. Une séquence donnée peut adopter plusieurs topologies en fonction de la nature et concentrations des cations, du pH, de sa concentration, de la température, de la présence de ligands, etc. La stabilité de ces structures est également fortement dépendante de ces facteurs. Si ce polymorphisme rend difficile l’évaluation des structures formées, il offre néanmoins la possibilité de cibler des gènes spécifiques, ou de concevoir des nanostructures.
Pour mieux comprendre le polymorphisme induit par les cations en particulier, nous avons lancé une étude visant à découvrir des séquences d’ADN dont les structures et stabilités présentent une très forte dépendance à la nature et à la concentration des cations, ce qui leur permet de fonctionner comme des commutateurs allostériques [13].
À cette occasion, j’ai encadré Marie Toulisse (IUT, Bordeaux ; désormais Responsable cellule approvisionnement à la SNCF) avec laquelle nous avons criblé de nombreuses séquences par dichroïsme circulaire. Après avoir identifié les séquences les plus prometteuses, nous avons caractérisé thermodynamiquement et cinétiquement l’influence des cations par de nombreuses techniques spectroscopiques (UV-vis, UV-melting, CD, RMN), spectrométriques (spectrométrie de masse native, mobilité ionique) et chromatographique (méthode SEC décrite ci-avant, Section 3.1).
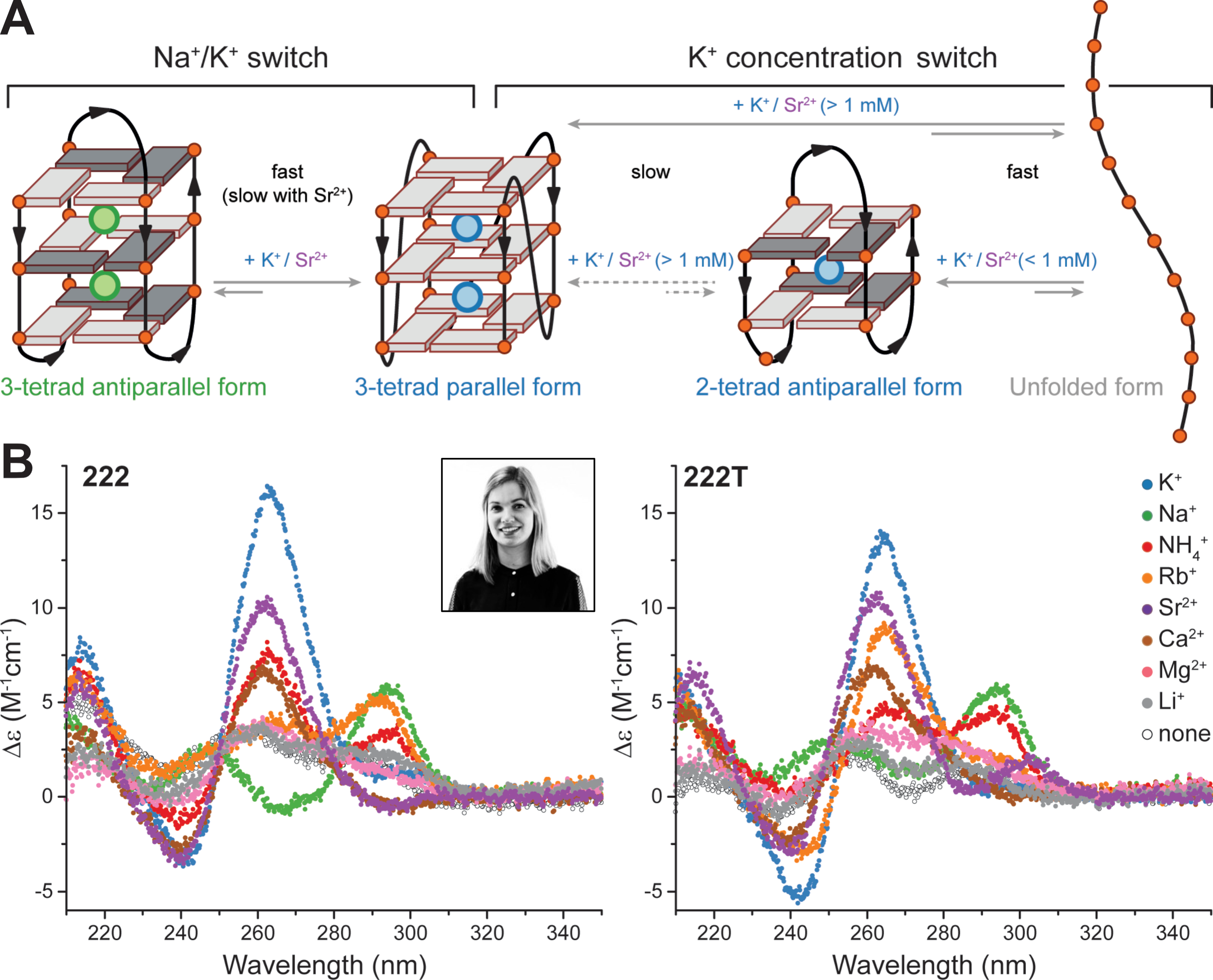
Le motif G3T2 s’est montré particulièrement prometteur et nous avons caractérisé en détails les séquences 222 (d[(G3T2)3G3]) et 222T (d[T(G3T2)3G3T]), qui fonctionnent comme des doubles commutateurs (Figure 3.4A). Le premier switch se fait entre un G4 à trois tétrades, antiparallèle et coordonnant du sodium et un G4 à trois tétrades, parallèle, et coordonnant du potassium. L’interconversion est déclenchée par l’ajout de petites quantités de potassium à un échantillon riche en sodium. La très grande différence de stabilité entre la forme Na+ et la forme K+ explique que de petites quantités de se dernier suffisent à une interconversion quantitative et instantanée (plus rapide que le temps mort expérimental) à température ambiante.
Le second switch se fait entre un G4 deux tétrades, antiparallèle, et un G4 trois tétrades, parallèle, par simple augmentation de la concentration en potassium. De manière générale, ces deux oligonucléotides se structurent de façon très dépendante à la nature du cation, ce qui en fait des modèles très intéressants à explorer (Figure 3.4B). J’ai confirmé et résumé la tendance globale de stabilisation des G4s en fonction de la nature du cation : Sr2+ > Ba2+ > K+ > Ca2+ > Na+ , NH4+ , Rb+ > Mg2+ > Li+ ≥ Cs+.
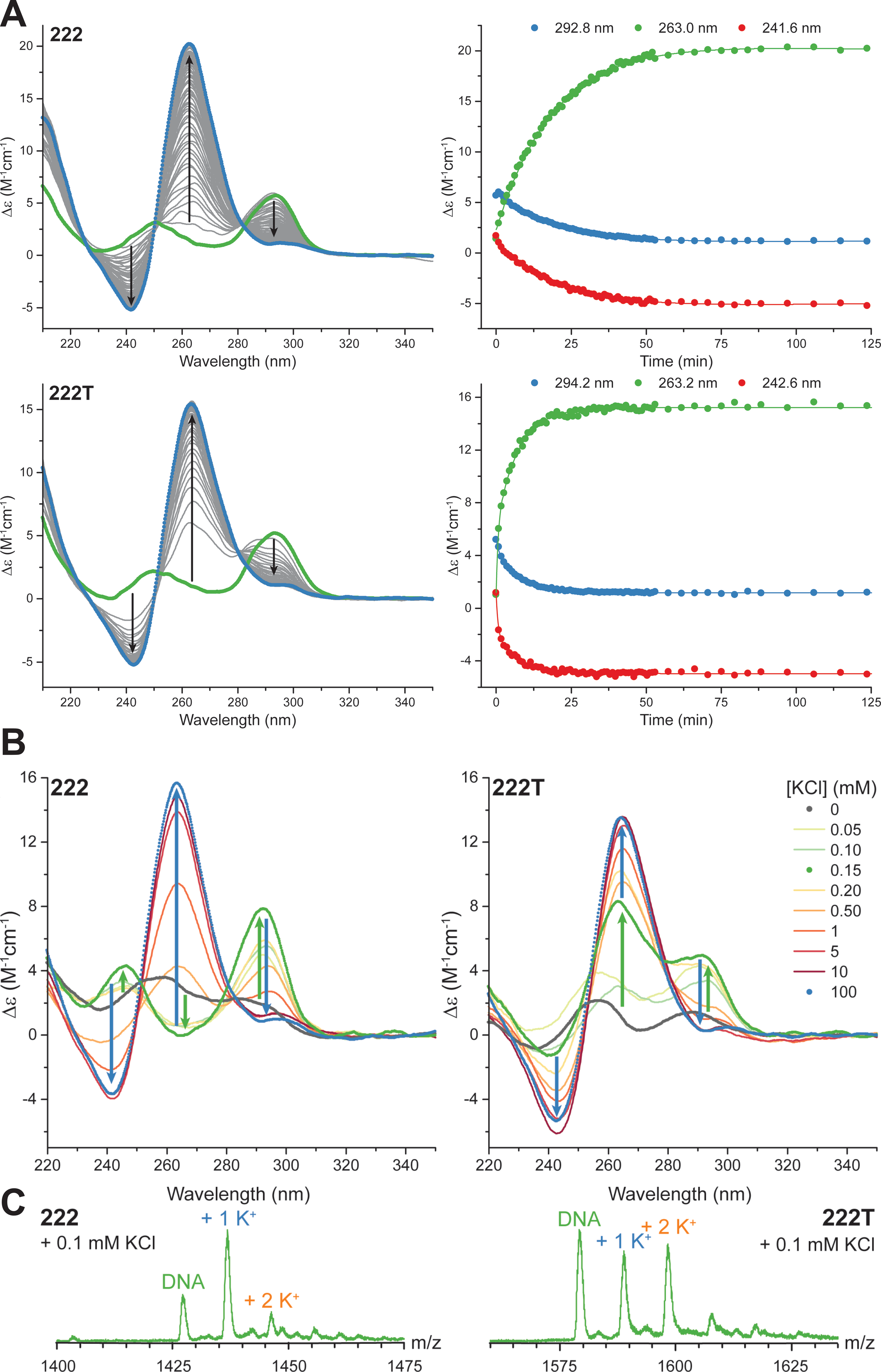
Les thymines en 5’ et 3’ de 222T, mais absente de 222, rendent l’interconversion plus rapide (cinétique en CD et UV-vis ; Figure 3.5A), la complexation du potassium plus coopérative (détermination de Kd par spectrométrie de masse native ; Figure 3.5B, C), et inhibe la dimérisation des G4s (détermination de stœchiométrie par MS native et SEC-HPLC, comme décrit ci-avant, Section 3.1). Ces commutateurs sont donc modulables avec des modifications discrètes et minimes.
Durant le criblage, nous avons mis en évidence que de nombreuses séquences riches en guanines adoptent le même comportement, même si souvent de façon moins marquée. Ces systèmes d’ADN présentent un intérêt fonctionnel (par exemple en tant que nanomachines), mais leur polyvalence structurelle en fait également un outil méthodologique précieux pour la compréhension de la thermodynamique et de la cinétique du repliement de l’ADN G4. La modulation facile de structures G4s en fonction des conditions expérimentales en fait également des modèles très pratiques pour le développement analytique. J’aurai d’ailleurs de nombreuses fois l’occasion d’utiliser 222T dans la suite de mes travaux, notamment à mon retour à l’IECB comme maître de conférences (voir le Chapitre 5, en particulier les Sections 5.2.1 et 5.3.1.1) [14–16].
Cette publication, ainsi qu’un chapitre de livre que j’ai écrit avec Valérie et Jean-Louis sur les interactions entre G4 et métaux alcalins [17], sont fréquemment cités par la communauté G4 (> 350 citations). Il s’agit d’un de mes papiers préférés car j’y fait appel à des techniques analytiques “basiques” mais permettant, avec un traitement des données ad hoc, de produire des résultats très significatifs.
3.3 Conclusion
Au cours de ce second postdoctorat, j’ai développé une approche par chromatographie d’exclusion stérique spécifiquement calibrée pour distinguer les polymorphismes structuraux des acides nucléiques (duplexes, i‑motifs, triplexes, G‑quadruplexes). Cette méthode a permis de caractériser les structures et la stœchiométrie de plus d’une centaine d’oligonucléotides. Par ailleurs, l’identification, par une combinaison de spectroscopies et spectrométrie de masse native de séquences capables de commuter entre topologies de G4 selon la nature ou la concentration des cations a révélé des “interrupteurs” moléculaires très sensibles. Ces travaux illustrent l’importance d’une approche analytique multi-technique pour étudier les structures non canoniques de l’ADN, dont l’important polymorphisme a été encore un peu plus mis en lumière.
Personnellement, c’est une évidence mais il convient tout de même de l’écrire, ce contrat postdoctoral jettera les bases de mon retour en 2017, mais pas uniquement. C’est également ces travaux qui m’ont réellement lancé dans le monde de la spectrométrie de masse (native) et m’ont amené au chapitre suivant de ma carrière, dans lequel il est beaucoup question de MS (mais pas native).