2 Postdoctorat à l’Université de Colombie Britannique
2.1 Contexte
J’ai rejoint l’équipe du Pr. David Perrin, au département de chimie de l’Université de Colombie Britannique (UBC) en septembre 2012 (Figure 2.1). Plusieurs thèmes de recherche y étaient développés, notamment la sélection d’aptamères d’ADN modifié avec des chaînes latérales d’acide aminés. L’objectif était de produire des DNAzymes pour cliver des mARN viraux, des sondes de métaux toxiques ou des aptamères se liant à la surface de cellules. D’autres projets incluaient la synthèse de molécules pour l’imagerie PET et la synthèse peptidique à des fins thérapeutiques. Plus trivialement, les étudiants et postdoctorants de l’équipe faisaient de la synthèse organique ou du SELEX. Et je n’ai donc fait ni l’un ni l’autre.
La majeure partie de l’information génétique se trouve à l’intérieur de la double hélice d’ADN, protégée par des interactions auto-complémentaires. Dans les années 80, Hélène et Dervan ont proposé l’hypothèse thérapeutique dite « antigène », qui implique le ciblage de séquences spécifiques d’ADN par des oligonucléotides complémentaires [1, 2]. La reconnaissance de l’ADN pour la thérapie génique, avec des oligonucléotides présentant par nature une sélectivité de séquence, a depuis été un axe majeur de recherche, notamment pour réguler les processus biologiques comme l’activation de la transcription de l’ADN [3]. Le déplacement de brin avec, par exemple, des PNA implique une séparation de la double hélice, remplaçant un set de paires de bases par un autre, ce qui n’est pas particulièrement favorable énergétiquement.
Les nucléosides Janus, proposés par Lehn, offre une approche de reconnaissance de l’ADN qui permet un gain énergétique en établissant simultanément des liaisons hydrogène avec les deux bases d’une paire, plutôt qu’en se substituant à un brin [4]. La formation de nouvelles liaisons hydrogène accroît la stabilité du complexe de l’ordre de 2 kcal.mol-1 par paire de bases. Les premiers nucléotides Janus étaient uniquement capables de reconnaître des paires TT ou TC [5, 6]. Le véritable intérêt en termes de reconnaissance de l’ADN réside dans la synthèse de nucléosides Janus qui présentent des donneurs de protons (D) et des accepteurs (A) disposés de manière à intercepter toutes les liaisons H que l’on trouve dans une paire de bases canonique.
Lorsque je suis arrivé dans l’équipe du Prof. Perrin, deux étudiants, Wenbo Liu et Abid Hasan, avaient débuté un travail de synthèse d’analogues de nucléotides Janus, donc disposant de deux faces distinctes possédant un réseau de sites donneurs et accepteurs de liaisons hydrogènes complémentaires de différentes bases. Dans ce contexte, j’ai apporté ma contribution pour la purification et la caractérisation biophysique et computationnelle d’oligonucléotides incorporant un nucléotide Janus AT, ce qui a permis de non seulement valider mais aussi optimiser le design de ces nucléotides modifiés [7, 8].
2.2 Synthèse, caractérisation et optimisation d’oligonucléotides incorporants des nucléotides bifaciaux
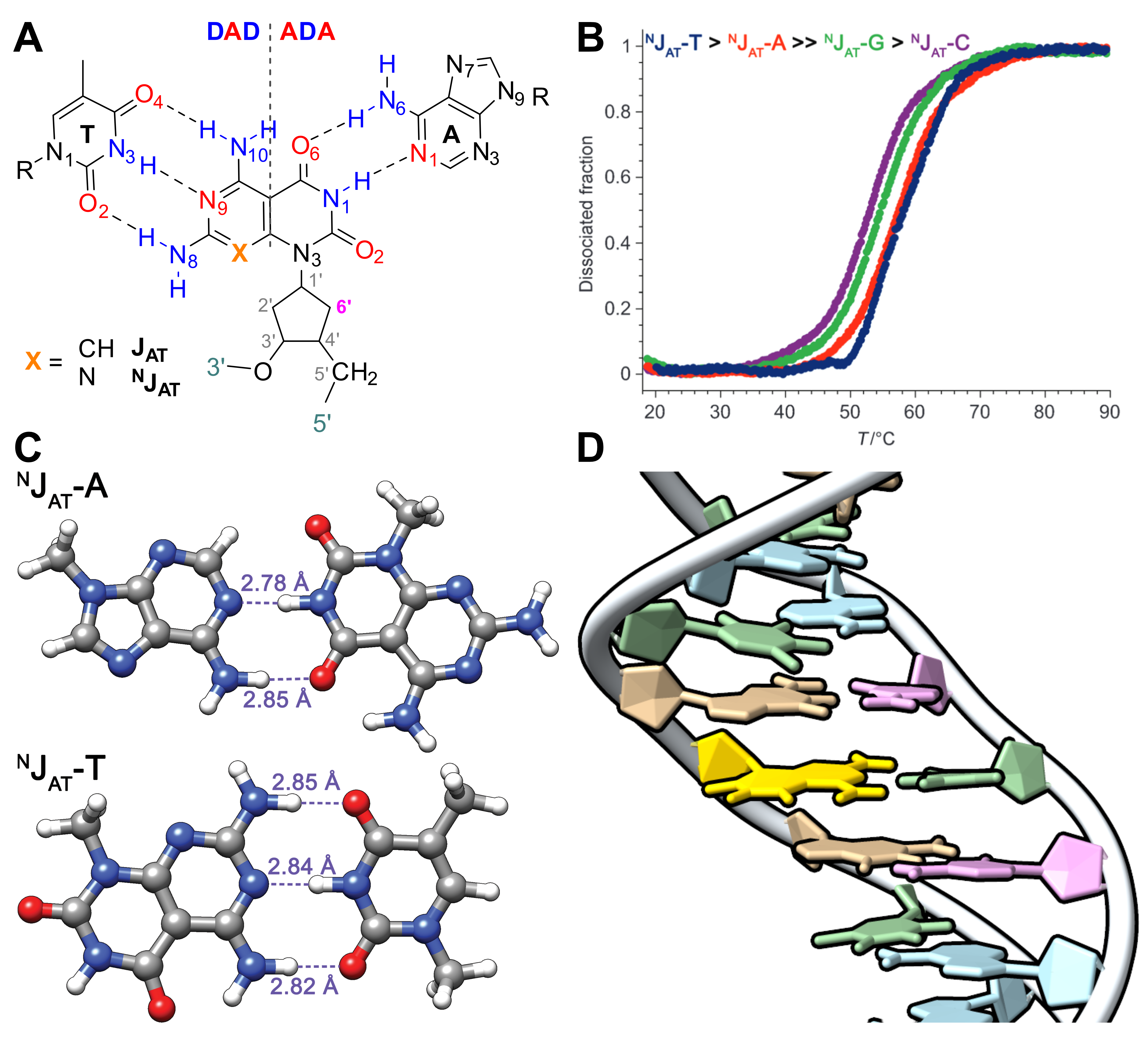
Nous avons donc développé une base Janus-AT « JAT », pouvant se lier simultanément à une adénine et une thymine (Figure 2.2A, X = CH) [7]. Le design du nucléotide est rationnel, avec une face ADA pouvant théoriquement formé trois liaisons hydrogène avec les thymines, et une face DAD pour hybridation avec les adénines. Pour éviter le clivage de la liaison N-glycosidique, un analogue carbocyclique du désoxyribose a été sélectionné. En outre, il a été montré que ce carbocycle améliore la stabilité des duplex et des triplex dans d’autres contextes [9]. Un oligonucléotide incorporant un JAT a été synthétisé à partir du phosphoramidate de JAT préparé par Wenbo et Abid. J’ai ensuite purifié cet oligonucléotide sur gel et vérifié l’identité par spectrométrie de masse MALDI-TOF.
J’ai démontré avec une première étude biophysique que ce nucléotide modifié permet bien l’hybridation avec une adénine ou une thymine dans le contexte d’un double brin d’ADN. Ainsi, en présence de l’oligonucléotide complémentaire (avec une adénine face au JAT), le duplex a bien été détecté en phase gazeuse par MALDI TOF. Le dichroïsme circulaire a confirmé la formation de duplexes en solution. Les expériences d’UV-melting ont démontrés que la paire JAT-T, mais pas la paire JAT-A, est plus stabilisante qu’une paire AT. En outre, les paires JAT-A ont générées des courbes dont les transitions particulièrement larges, avec une Tm inférieure aux paires AT mais supérieure à un mésappariement. J’ai également pris soin de montrer que JAT est spécifique de A et T, en mesurant les Tm avec C et G, ces deux nucléotides entrainant une baisse de la température de dénaturation de l’ordre de 10°C.
Pour tenter d’expliquer ces résultats prometteurs mais pas optimaux, j’ai élargi mes horizons en me tournant vers la chimie computationnelle. Ainsi, par calculs DFT (BP86/TZ2P) sur des paires de nucléotides isolés, j’ai obtenu des résultats cohérents avec la formation de paires de bases, là aussi suggérant que JAT-T est plus stable que JAT-T. En revanche, la répulsion importante entre groupes carbonyles ne permet pas, comme attendu, l’hybridation à G ou C.
J’ai ensuite réalisé des simulations de dynamiques moléculaires de doubles hélices incorporant un JAT, dans un champ de force AMBER avec solvatation implicite. La double hélice est restée stable pendant la simulation de deux nanosecondes.1 Lorsqu’il est associé à A, JAT est cependant contraint dans une conformation qui ne maximise pas le \(\pi\)-stacking, ce que nous avions pensé pouvoir être à l’origine de la stabilité inférieure de ce duplex.
1 Ces simulations peuvent paraître très courtes au lecteur, en particulier au regard de mes travaux actuels, mais il s’agit d’un petit miracle de la science si l’on considère que ces calculs ont été réalisés sur mon ordinateur portable - qui s’est mû en chauffage d’appoint pendant plusieurs semaines.
Nous avons finalement fait l’hypothèse que le problème était plus… basique. Plus spécifiquement, nous avons suggéré que le comportement anormal de la paire JAT-A pouvait être dû à la protonation partielle à pH physiologique de l’azote N9, pour lequel j’avais calculé \(pKa \approx 7.5\). Cela conduirait à un mélange en équilibre dynamique la base neutre qui peut s’apparier et sa forme protonée pour laquelle l’appariement est peu probable.
Nous avons donc synthétisé un nucléotide de seconde génération « NJAT », dans lequel l’inclusion d’un azote endocyclique rend l’hétérocycle considérablement moins basique, minimisant ainsi le potentiel de protonation en une espèce cationique incapable de s’apparier (Figure 2.2A, Figure 2.3) [8]. Les expériences de melting ont cette fois montré que NJAT produit des transitions nettes, et discrimine efficacement A/T par rapport à G/C (Figure 2.2B). Les expériences de dichroïsme circulaire et dynamiques moléculaires ont confirmé qu’il y a peu ou pas de modification de la structure secondaire pour les duplex formés (Figure 2.2C, D). NJAT produit donc des paires de bases stables avec A et T et est donc un candidat prometteur pour développer des stratégies de ciblage de séquences d’ADN.
Ces travaux postdoctoraux ont été possibles grâce à l’obtention d’une Bourse Banting, délivrée par le Conseil de recherches en sciences naturelles et en génie du Canada. Pendant ce séjour, j’ai co-encadré Wenbo Liu (Master) et Abid Hasan (Master), tous les deux participant à la synthèse de nucléotides Janus.
2.3 Conclusion
L’objectif principal de ce séjour postdoctoral était d’explorer les nucléotides Janus, capables de reconnaître simultanément deux bases complémentaires. Suite à la synthèse du dérivés JAT, j’ai démontré sa capacité à former spécifiquement des paires de bases avec A et T, puis rationalisé le design d’une base optimisée NJAT, grâce à une combinaison de biophysique et chimie computationnelle.
D’un point de vue personnel, cette expérience m’a conforté dans l’orientation plus analytique de ma carrière et m’a beaucoup appris, scientifiquement et humainement. J’ai publié mes premiers travaux de chimie computationnelle (Figure 2.3), et élargi mes connaissances en biochimie des oligonucléotides et en SELEX, même si cela ne s’est pas traduit par des publications à court terme. En particulier, j’ai participé à des travaux de sélection d’aptamères possédants des bases modifiées, capables de cliver de l’ADN et de l’ARN en conditions physiologiques. Ces travaux m’ont finalement mené à l’analyse de la cinétique d’incorporation de nucléotides modifiés comme substrats de polymérases obtenues par évolution dirigée [10].