1 Doctorat à l’Institut Curie/CNRS UMR176/Université Paris XI
1.1 Contexte
J’ai effectué mon doctorat à l’Institut Curie, au sein de l’UMR176 du CNRS, rattachée à l’Université Paris XI, grâce à une bourse de docteur ingénieur du CNRS (co-financée par l’Institut Curie). Marie-Paule Teulade-Fichou (Figure 1.1), directrice de l’unité, dirige mes travaux de thèse centrés sur l’étude de l’interaction de petites molécules avec les acides nucléiques G-quadruplex (G4). Nous retrouverons cette famille de structures secondaires non-canoniques de l’ADN et l’ARN tout au long de ce mémoire, et quelques éléments de contexte sont donc nécessaires. Par soucis de concision, ces structures ne sont pas décrites ci-dessous avec le niveau de détails dont je peux habituellement faire preuve. Le lecteur curieux, mais pressé, peut se référer à un guide du G4 concis que j’ai mis en ligne. Pour une description plus exhaustive des structures secondaires d’ADN et leurs interactions non-covalentes, le lecteur peut se référer à la section 2 de la référence [1].
1.1.1 Les G-Quadruplexes
Les G-Quadruplexes (G4) sont des quadruples hélices d’acide nucléique, formées par un (intramoléculaire), deux (bimoléculaire) ou quatre (tétramoléculaire) brins d’acide nucléique riches en guanines et, de préférence, pauvres en cytosines [2, 3]. Les G4 sont impliqués dans un certain nombre de processus biologiques critiques (au niveau de l’ADN et de l’ARN), ce qui en fait des cibles thérapeutiques prometteuses (par exemple, régulation des gènes, inhibition de la télomérase, lésions de l’ADN) [4–7]. Ils sont parfois formés par des aptamères et peuvent être utilisés en nanotechnologie (Figure 1.2) [8, 9].
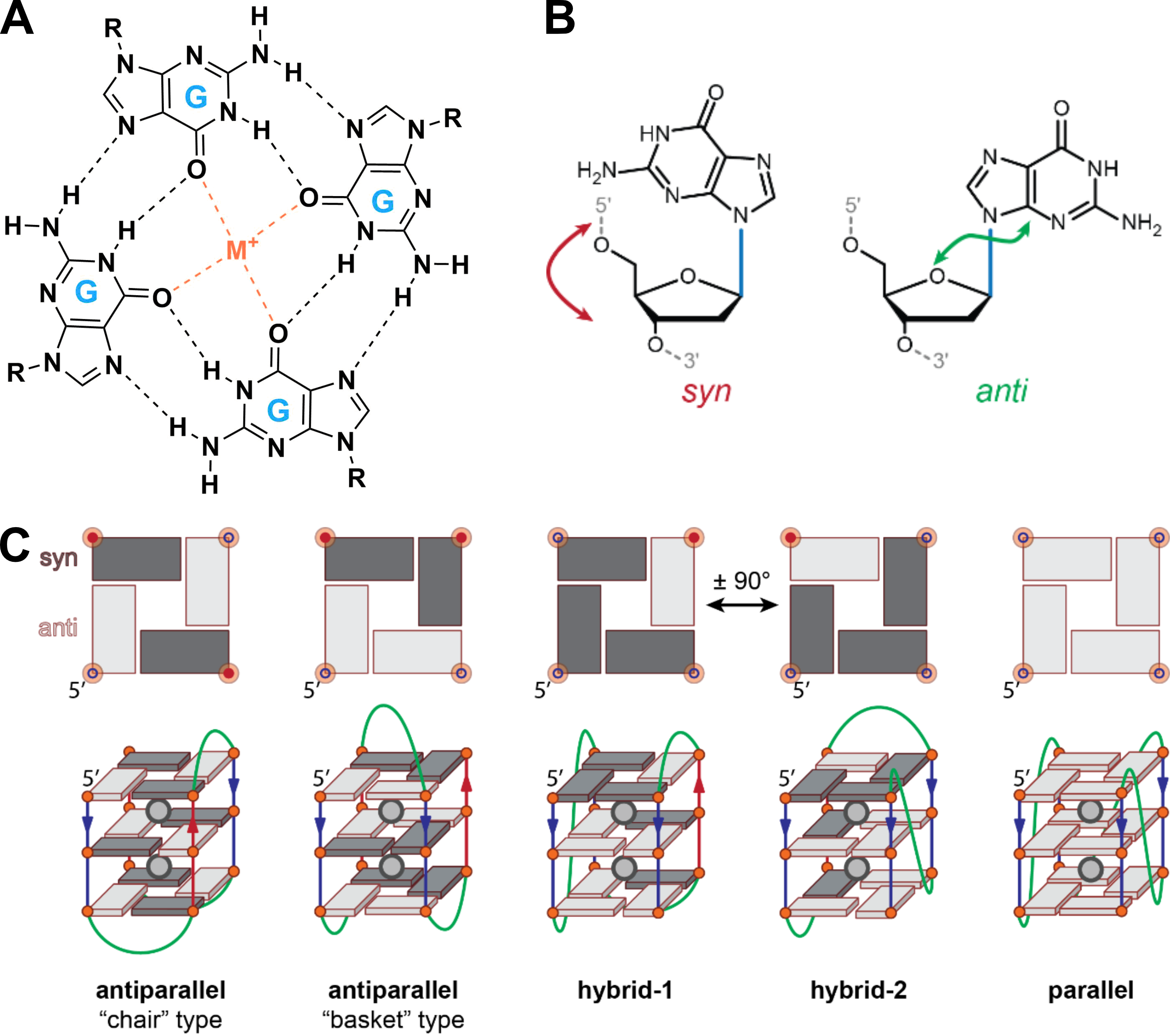
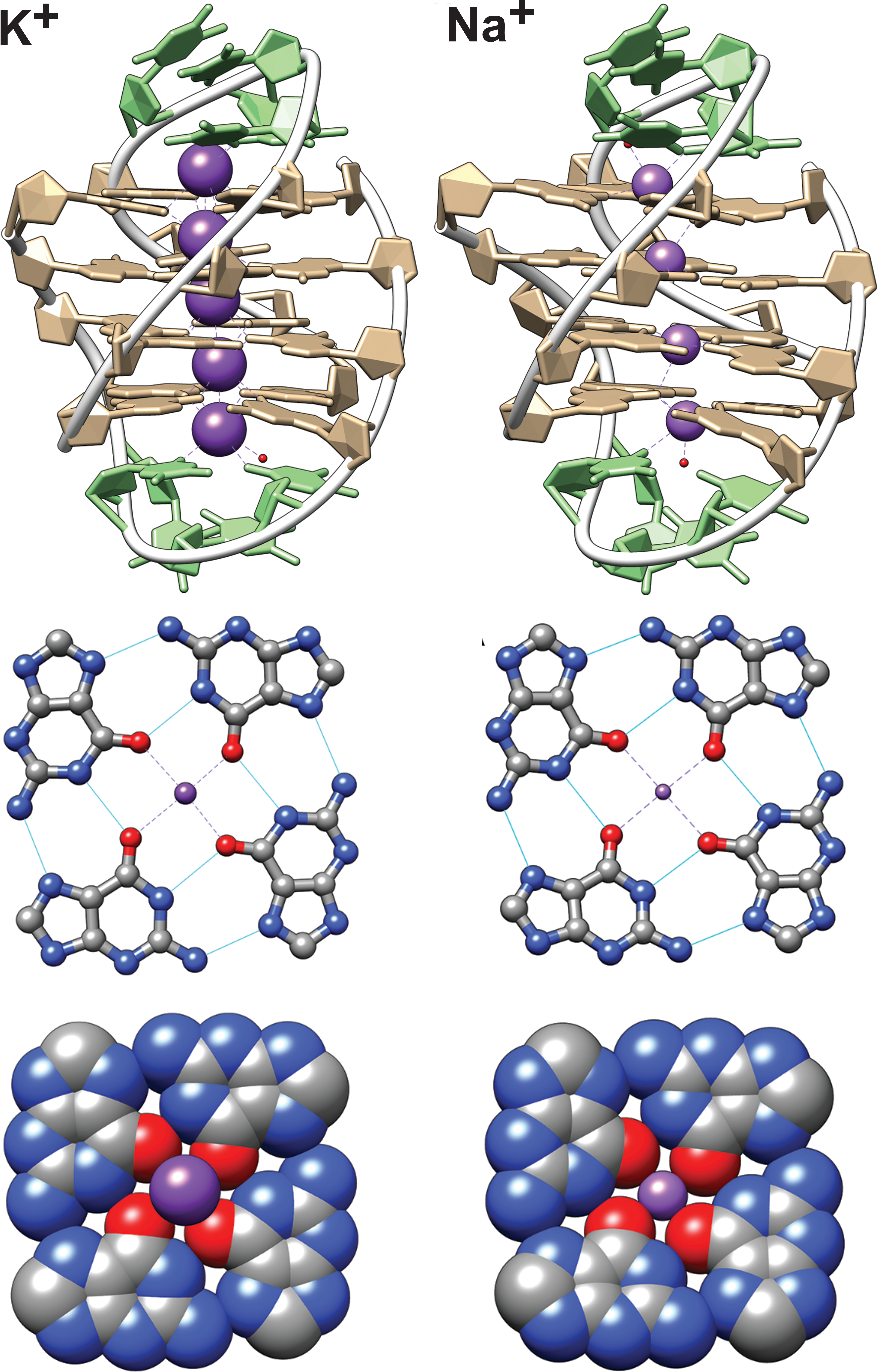
Les G4 sont formés par un empilement d’au moins deux G-quartets, également appelés G-tétrades (Figure 1.3A). Une tétrade est elle-même composée de quatre guanines associées par huit liaisons hydrogène sur leurs faces de Watson-Crick et de Hoogsteen. Un cation métallique (typiquement K+ ou Na+) est coordonné par le O6 de huit guanines appartenant à deux tétrades G empilées consécutivement (Figure 1.4) [10]. Par conséquent, un G-quadruplex contenant n G-quartets coordonne généralement \(n-1\) cations. Le rayon ionique influence grandement la capacité d’un cation à promouvoir la formation des G-quadruplexes, ce que j’exporerai plus tard dans mon parcours (Section 3.2) [10–13].
Dans un G4, les guanines impliquées dans les G-quartets constituent les G-tracts. Les nucléotides des termini 5’ et 3’, positionnés avant le premier ou après le dernier G-quartet, sont appelés flanking sequences. Les boucles reliant les G-tracts peuvent adopter différentes géométries, principalement la propeller (reliant des guanines de G-tracts adjacents mais de quartets opposés), latérale (G-tracts adjacents et même quartet), et diagonale (G-tracts diagonalement opposés et même quartet) [16].
Les G4 sont particulièrement polymorphes ; certaines séquences peuvent adopter des conformations distinctes, coexistant parfois en équilibre [17, 18]. Les résidus des termini et des boucles peuvent favoriser des conformations spécifiques en formant des paires de bases avec d’autres résidus de termini ou des boucles et en s’empilant sur des tétrades externes [19–21]. En règle générale, les séquences avec des boucles plus courtes ont tendance à adopter une géométrie propeller et à produire des G4s plus stables que celles avec des boucles longues [22].
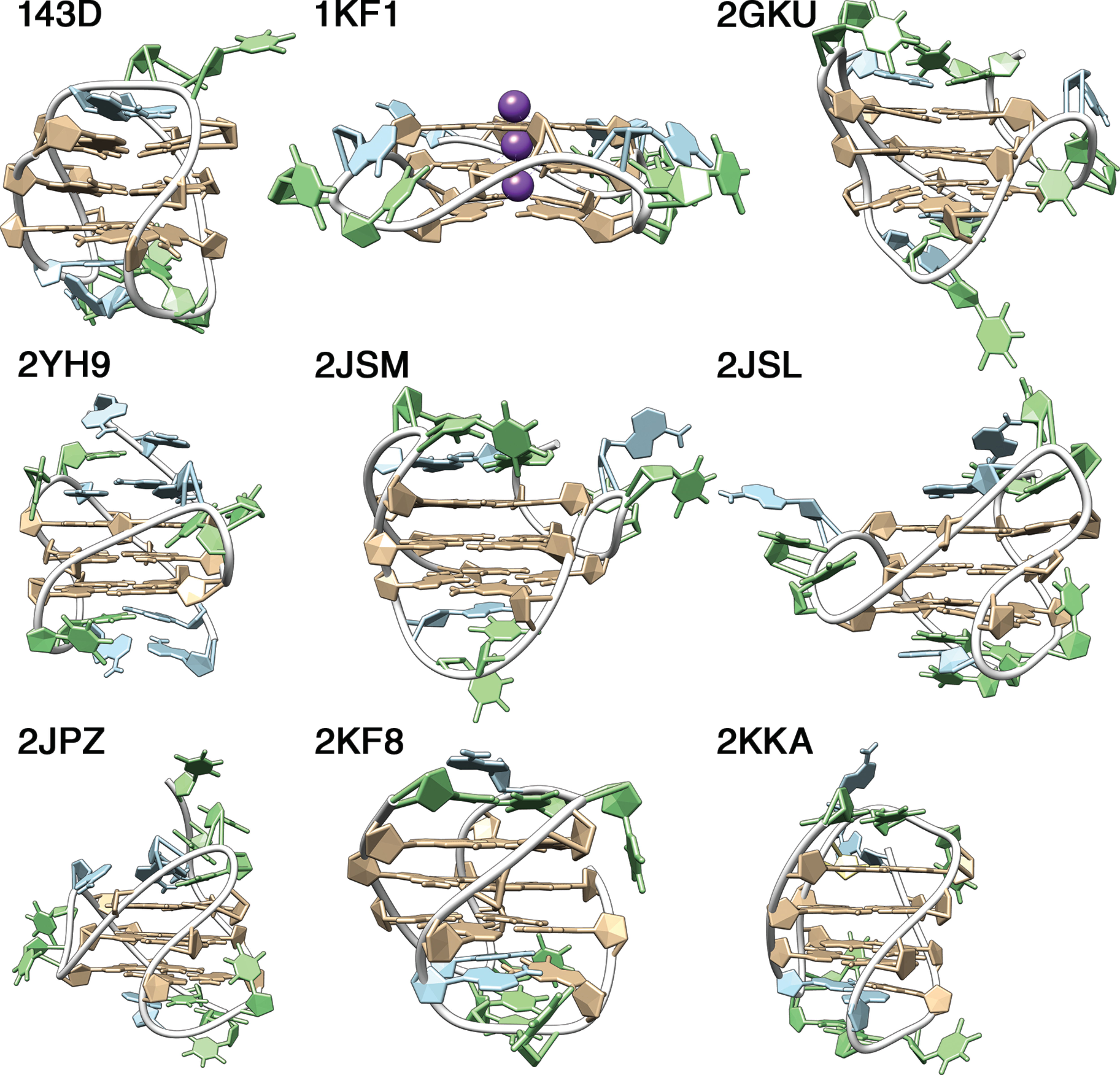
L’attention particulière portée à la séquence télomérique humaine est en partie historique, avec un certain nombre d’études fondamentales parues à la fin des années 80 et au début des années 90 [23–25], en partie thérapeutique [26–28], mais découle également de son polymorphisme particulièrement impressionnant pour une simple répétition des bases GGGTTA. On pourrait écrire des pages sur ce polymorphisme, ce dont je vous ferai grâce ici puisque je l’ai déjà fait par ailleurs [10], et que la Figure 1.5 illustre plutôt bien cette diversité structurale.
Ce qu’il faut préciser en revanche, c’est que ce polymorphisme est loin de se limiter à la seule séquence télomérique humaine. Étant donnée cette polymorphie intrinsèque des G4s, ils ont rapidement été classée dans des familles partageant une même topologie. Celle-ci est généralement définie par les orientations relatives des G-tracts et/ou des angles de liaison glycosidique des guanines (syn ou anti ; Figure 1.3B). La topologie est parallèle lorsque tous les tracts sont orientés dans la même direction (toutes les guanines empilées ont un angle de liaison glycosidique en anti), antiparallèle lorsque deux brins sont orientés dans la direction opposée aux deux autres (hétéro-empilement de guanines syn et anti) et hybride lorsqu’un brin est orienté dans la direction opposée aux trois autres (mélange d’homo- et d’hétéro-empilement) (Figure 1.3C) [16]. Les topologies antiparallèles où les brins de direction opposée sont latéralement (type “chaise”, toutes les boucles sont latérales) ou diagonalement (type “panier”, une boucle diagonale et deux latérales) opposés ne sont pas équivalentes. On notera que la topologie hybride a été introduit tardivement (en 2006 [37]), et par conséquent une grande partie de la littérature antérieure ne mentionne que les topologies parallèles et antiparallèles d’une façon qui peut maintenant nous paraître erronée [38].
1.1.2 Les ligands de G-Quadruplexes
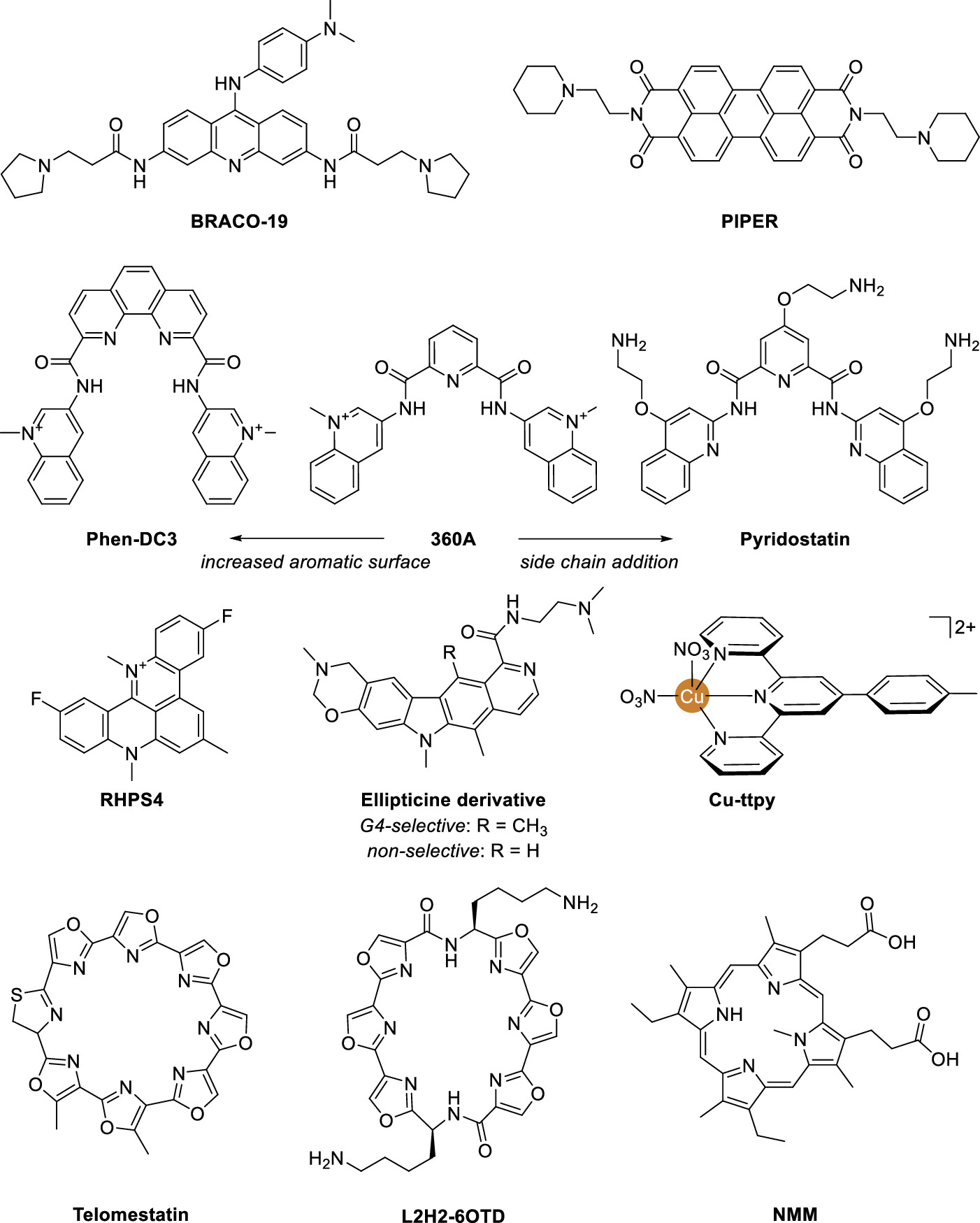
La découverte de molécules capables de se lier sélectivement à ces structures est une piste thérapeutique prometteuse [39–41]. Les premières générations de ligands de G4s a été fortement inspirée par les intercalants d’ADN duplex. Ainsi, un des premiers ligand de G4 connu est RHPS4, qui est un ligand d’ADN duplex (Figure 1.6) [42]. Compte tenu de la différence de taille et de forme entre les tétrades et les paires de bases, l’augmentation de l’affinité et de la spécificité peut reposer sur une simple augmentation de la surface aromatique afin d’optimiser son chevauchement avec tétrades tout en empêchant son intercalation. Le PhenDC3, qui présente une excellent affinité pour les G4s et une sélectivité élevée contre l’ADN duplex, est un bon exemple d’optimisation rationnelle de la taille et de la forme du cœur aromatique [43]. L’ajout de chaînes latérales protonables et l’optimisation de leur nature et de leur longueur peuvent également contribuer favorablement à l’interaction, moduler la sélectivité et améliorer la solubilité dans l’eau [44–49]. Un exemple précurseur est BRACO-19, une acridine 3,6,9-trisubstituée dont les pyrrolidines des chaînes latérales permettent des interactions favorables avec les sillons, tandis que le substituant 9-anilino se positionne dans un troisième sillon [42, 44, 50]. De même, la pyridostatine est un dérivé de la 360A dans lequel trois chaînes latérales ont été ajoutées et les quinolines ne sont plus N-méthylées [51].
1.2 Orientations des travaux de thèse
Mon sujet de thèse était initialement uniquement dédié à la synthèse de complexes métalliques comme ligands de l’ADN quadruplexe. Il devait venir continuer (une partie) du travail d’une précédente doctorante, le Dr. Hélène Bertrand, qui a produit plusieurs articles sur le sujet [52–55]. Comme Hélène part au moment où j’arrive, c’est David Monchaud, chargé de recherche CNRS au laboratoire, qui me prend sous son aile et m’inculque tout ce que j’ai besoin de savoir sur l’ADN, et plus, le jour de mon arrivée. L’ampleur de la tâche et la force du café de David me donnera des palpitations. Il me forme également sur les techniques spectroscopiques que je serai amené à utiliser [56–58], ce qui sera d’une grande importance.
1 Généralement par des personnes qui travaillent toujours dans le domaine.
Dans les faits, au début de mes travaux de thèse (en octobre 2008), de nombreuses molécules ont déjà été publiées, dont certaines - comme 360A, PhenDC3 ou Cu-ttpy - dans mon laboratoire de thèse, avec des sélectivités significatives en faveur des G4s (sans pour autant discriminer efficacement différentes topologies) [43, 53]. J’entends d’ailleurs ici et là qu’on a fait le tour de la question, que les G4s sont un effet de mode et que dans 5 ans on en parlera plus, comme les triplexes.1 Il s’agit d’un élément de contexte important, car il va naturellement m’imposer un défi scientifique qui n’était pas explicitement prévu : il ne s’agit pas de simplement synthétiser de petites molécules qui se lient aux G4s mais de découvrir des ligands à valeur ajoutée. Je présente dans ce chapitre mes tentatives de ligands covalents, fluorescents, biotinylés, et qui discriminent des topologies de G4s, mais nous verrons que j’y reviendrai bien plus tard (Section 5.3) [59]. J’utilise ici le terme découverte car ces ligands ne sont pas tous le fruit du design rationnel. Pour obtenir des ligands “intéressants”, je vais aussi m’intéresser au criblage de petites molécules, ce qui va m’amener à développer des techniques analytiques ad hoc. Cela constituera un point de bascule dans mon parcours et enterrera ma carrière de chimiste organicien - ce qui n’est pas une grande perte pour la recherche. Cela n’aurait pas été possible sans l’apport inestimable de Florian Hamon (Figure 1.1 ; maintenant Head of New Drug Application group, chez Curium Pharma), qui a rejoint l’équipe comme postdoctorant environ un an après le début de mon doctorat. C’est lui qui prendra en charge le gros du travail de synthèse, étant de son côté peu attiré par les aspects analytiques. Cette complémentarité entre nos profils et envies a sans aucun doute joué un rôle majeur pour ma thèse et la suite de ma carrière.
En résumé, deux axes de travail interconnectés se sont donc dégagés :
Synthèses de nouveaux ligands organiques et inorganiques d’ADN quadruplexe,
Développement de méthodes analytiques pour l’analyse d’interactions entre ligands et acides nucléiques,
L’application des méthodes de l’axe 2 permet de caractériser les ligands préparés a l’axe 1, mais aussi d’en découvrir de nouveaux par criblage de chimiothèques.
1.3 Axe 1. Synthèse de nouveaux ligands organiques et inorganiques de haute affinité et sélectivité
Comme évoqué ci-dessus, les ligands de G4s publiés au début des années 2000 sont des molécules hétérocycliques planes, inspirés des intercalants de l’ADN duplex. Leur surface aromatique est généralement étendue par rapport à ces derniers pour maximiser le \(\pi\)-stacking sur les G-quartets tout en empêchant l’intercalation dans les duplexes [1, 60]. Plusieurs possèdent donc de bonnes voire excellentes sélectivités en faveur des G4s, mais pas entre plusieurs G4s (voir Section 1.4.1.2.1). Il existe également à l’époque peu de ligands possédant des fonctions supplémentaires que celle de se lier aux G4s. Une part importante de mes travaux de thèse a donc été la synthèse et caractérisation de nouveaux ligands organiques et inorganiques de haute affinité et sélectivité, possédants un ou plusieurs modes d’interaction et/ou des fonctionnalités d’intérêt (fluorescence pour la détection de G4s, couplés à de la biotine pour des expériences de pull down, etc.). J’ai ainsi synthétisé, ou participé à la synthèse, de plusieurs séries de ligands que je présente brièvement ci-après.
1.3.1 Complexes organométalliques comme ligands covalents et non-covalents des G4s
Ce projet est fondamental dans ma carrière, et en un sens forme le trait d’union entre ma formation initiale et certains de mes projets actuels. Il s’agit de mon projet initial de thèse, celui qui était présenté sur l’annonce à laquelle j’ai répondu, et sur lequel j’ai passé trois ans - mais pas à temps plein comme on le verra ci-après.
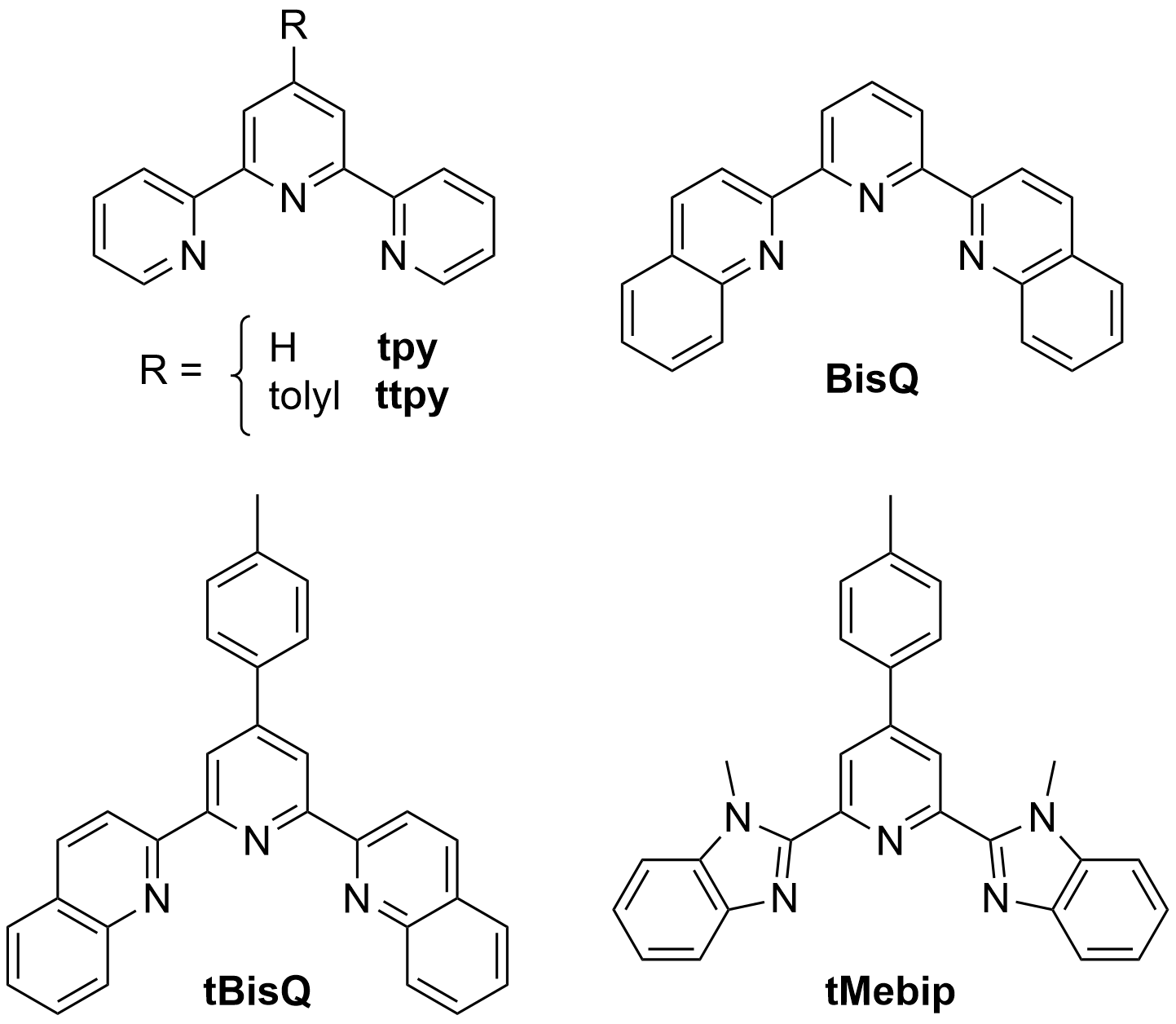
Le projet reprend là où ce sont arrêtés (l’excellente) doctorante qui m’a précédé, le Dr. Hélène Bertrand, désormais professeure au Laboratoire des BioMolécules (ENS, Paris), et Eric Talbot, un étudiant de Master ayant comme moi étudié à l’École Supérieure de Chimie Organique et Minérale, et maintenant Head of Synthetic Drug Design chez Isomorphic Labs (Londres, RU)2. Très brièvement, plusieurs complexes métalliques, particulièrement de platine et de cuivre, avec des ligands hétérocycliques (terpyridine: tpy, tolyl-terpyridine: ttpy, dibenzoterpyridine: BisQ ; Figure 1.7) ont été synthétisés et montre des affinités et sélectivités prometteuses pour les G4s [52, 53, 55]. En particulier, il apparaît que:
2 À ne pas confondre avec l’artiste qui a travaillé sur les Tortues Ninja
Les complexes de platine peuvent platiner l’ADN, ce qui est fréquemment exploité dans le cadre de thérapies anticancéreuses (inhibition de la réplication, altération des mécanismes de réparation) [61] ;
Les ligands apicaux des complexes de cuivre(II), rendant la molécule non-plane, pourraient jouer un rôle dans cette sélectivité: ils empêcheraient l’intercalation dans les duplexes et pourraient également empêcher le stacking sur des G4s dont les tétrades sont coiffées de boucles, entraînant une gêne stérique [53].
Ce projet sied le jeune doctorant que je suis car je sors d’un stage de M2 recherche centré sur la synthèse de molécules organométalliques, et il se situe à l’interface avec la biologie, ce qui m’attire depuis mon stage ingénieur chez Sanofi Aventis. Il sera aussi le projet qui m’amènera à collaborer avec Valérie Gabelica et Frédéric Rosu, ce qui conditionnera beaucoup la suite des évènements.
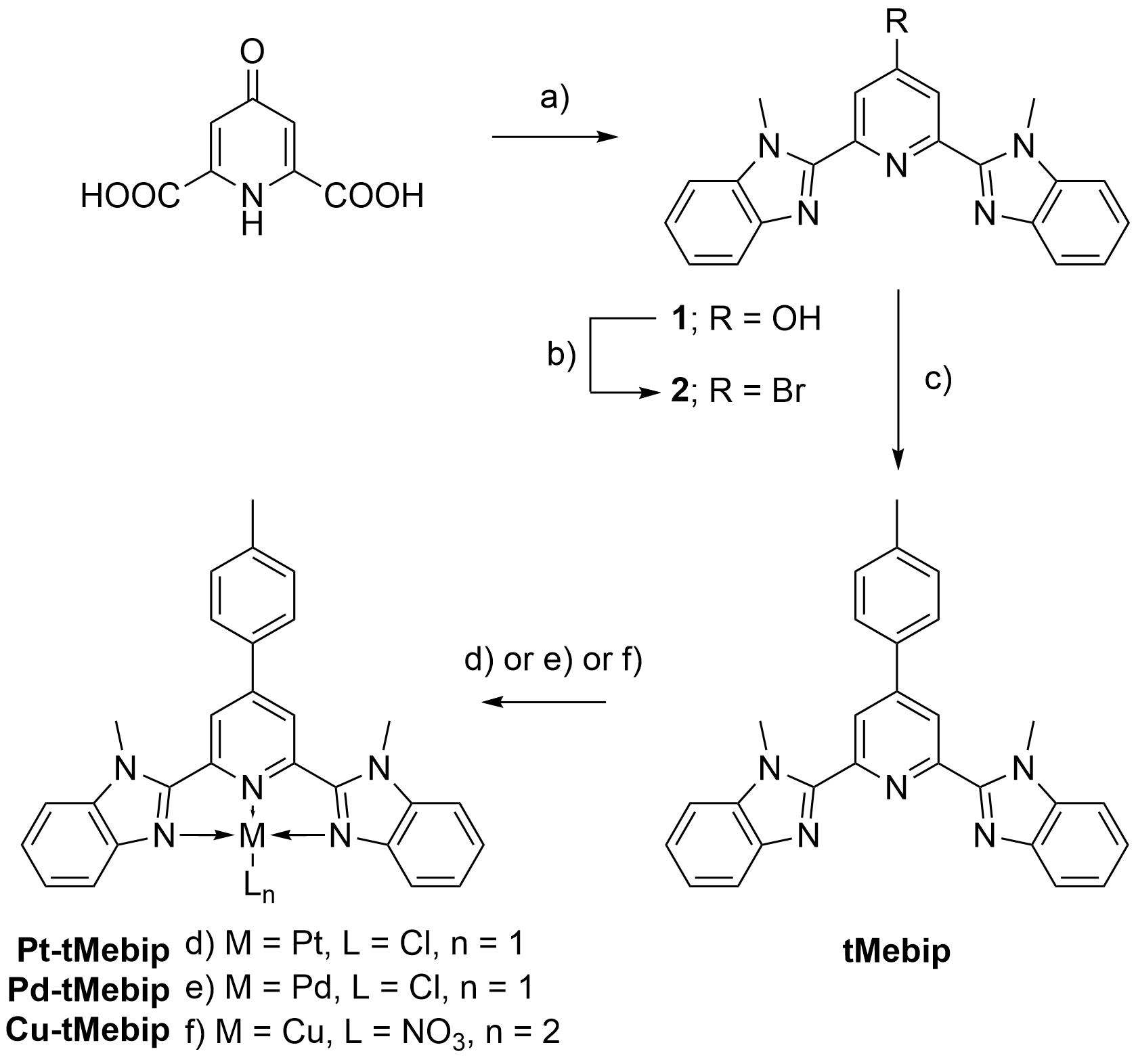
Une série de ligands hétérocycliques, azotés, tridendates commerciaux (terpyridine, tolylterpyridine) ou que j’ai synthétisés (BisQ, tBisQ, tMebip ; Figure 1.7) a été utilisée pour obtenir quinze complexes de palladium (II), platine (II) et cuivre (II) [62]. La synthèse de BisQ a déjà été décrite au laboratoire [52], en revanche celles de tBisQ et tMebip sont originales.
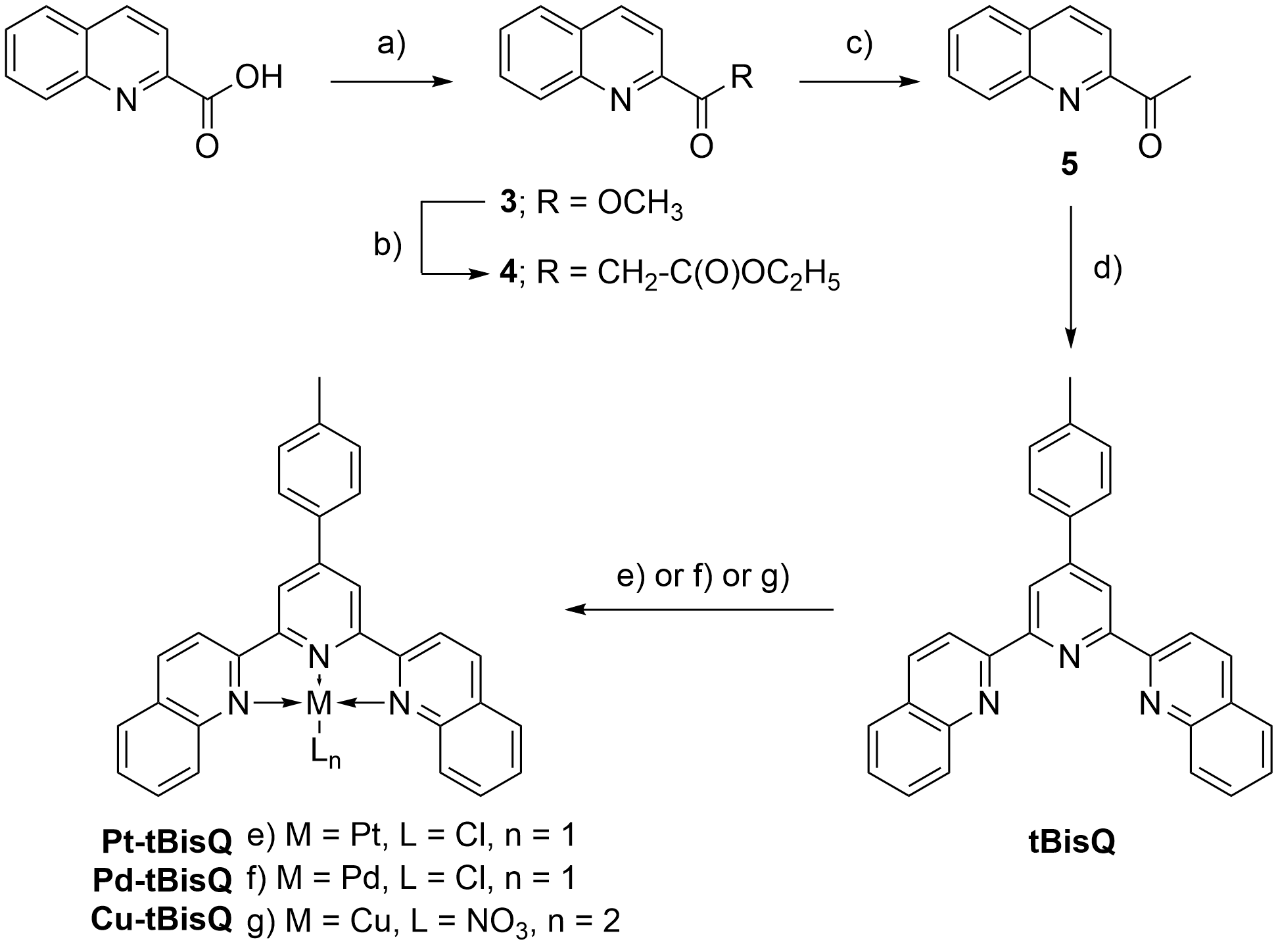
Pour tBisQ, l’intermédiaire clé, la 2-acétylquinoline, a été obtenu par estérification de l’acide quinaldique, suivie d’une condensation de Claisen, puis d’une étape de saponification/décarboxylation (Figure 1.8). Le ligand a finalement été obtenu avec un rendement de 54% par une double réaction de Kröhnke, en utilisant une double condensation avec de l’ammoniaque sur la 2-acétylquinoline, l’acétate d’ammonium et le 4-méthylbenzaldéhyde dans un tube scellé à 100°C pendant 24 heures [63]. Le rendement final fut de 27%.
Le tMebip a été préparé en trois étapes à partir de l’acide chélidamique, avec un rendement global de 45% (Figure 1.9). La formation des benzimidazoles a été obtenue par cyclisation thermique de l’acide chélidamique et de la N-méthyl-1,2-phénylènediamine en présence d’acide polyphosphorique, qui a été utilisé comme solvant [64]. La position 4 de la pyridine a ensuite été bromée par l’oxybromure de phosphore [65], ce qui a permis d’introduire le groupe tolyle par un couplage de Suzuki [66]. Le produit final a été facilement obtenu car il précipite à température ambiante dans le mélange THF/eau utilisé comme solvant. Cette synthèse a être réalisée à l’échelle du gramme et nécessite peu d’étapes de purification.
Les complexes de palladium, de platine et de cuivre de BisQ, tBisQ et tMebip ont été préparés en adaptant les procédures décrites dans la littérature [53, 67–69]. Ces complexes ont été obtenus avec des rendements inférieurs à ceux obtenus avec tpy et ttpy, probablement en raison de leur plus grande surface aromatique, qui délocalise les doublets non liant de l’azote. Ce sont d’ailleurs mes difficultés à produire le complexe Pt(II)-tMebip qui m’ont amené à préparer le complexe de Pd(II), cet ion étant plus réactif. Il ne s’agit alors que du deuxième exemple publié de complexes de palladium comme ligand de G4s, le premier venant de l’équipe de Ramon Vilar [70].
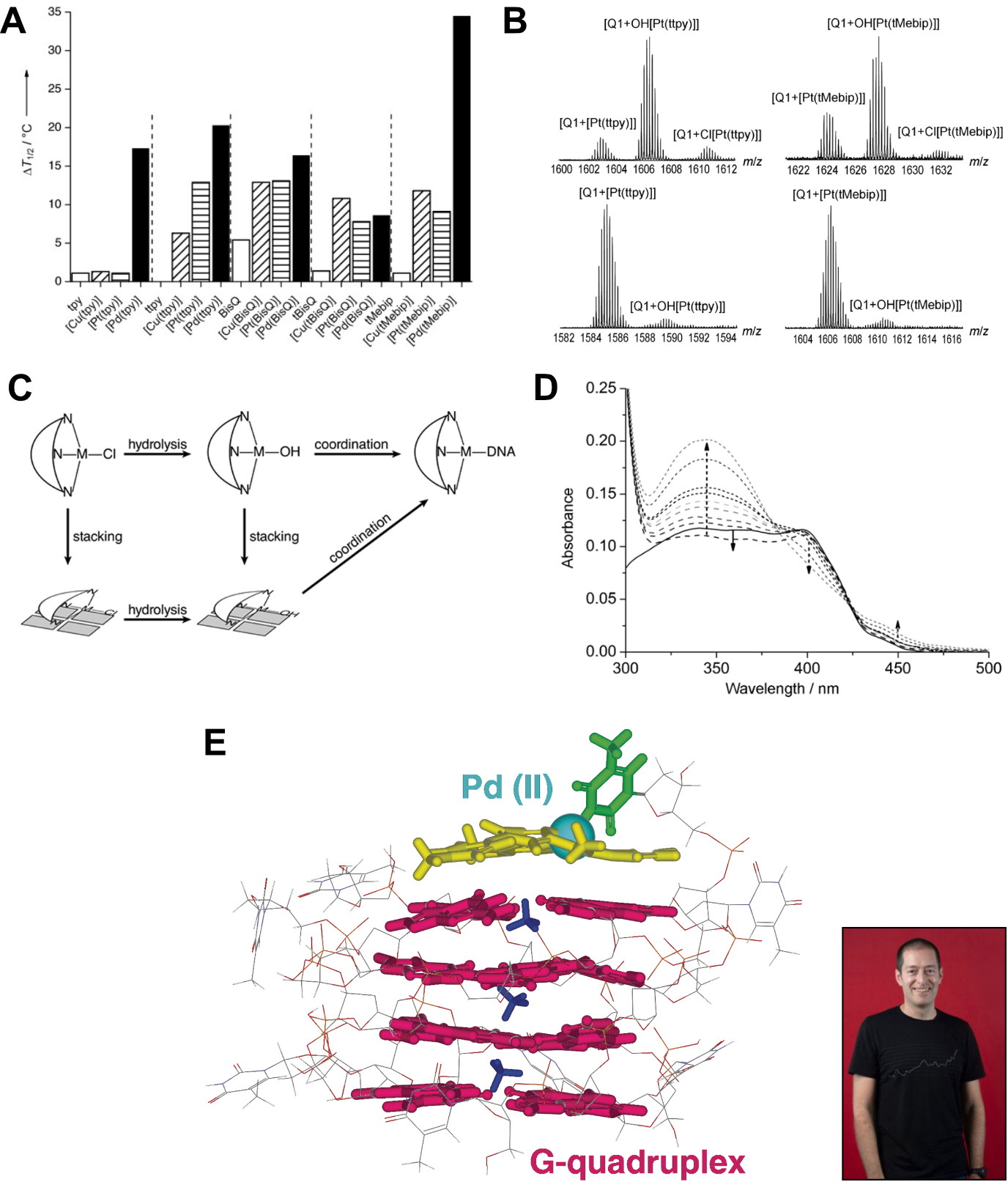
Nous avons observé une augmentation de la température de dénaturation des G4s impressionnante en présence des complexes de palladium, qui suggère une interaction forte (collaboration avec Aurore Guédin et Jean-Louis Mergny, ARNA, Bordeaux) (Figure 1.10A). Les complexes de platine et de cuivre donnent des stabilisations moindres, montrant l’importance de la nature du métal. La structure du ligand organique exerce également une influence, les surfaces aromatiques plus larges favorisant le stacking.
L’effet particulier des complexes de palladium s’explique par une coordination quantitative du cation métallique aux bases de l’ADN, que nous avons mis en évidence par spectrométrie de masse native avec Frédéric Rosu et Valérie Gabelica, alors à l’université de Liège, en Belgique (Figure 1.10B). Trois espèces ont été distinguées résultant de i) la coordination directe entre l’ADN et le complexe métallique, et du \(\pi\)-stacking non covalents entre l’ADN et les ii) formes chloré ou iii) aqua du complexe métallique (Figure 1.10C). Pour les complexes de platine, la forme principale liée à l’ADN est la forme aqua (Cl est remplacé par OH), mais quelques complexes chlorés associés à l’ADN sont encore observés. Le complexe directement coordonné à l’ADN, pour lequel un résidu de l’ADN occupe le quatrième site de coordination, est mineur pour les complexes de platine, mais très largement prédominant pour les complexes de palladium (quantitatif à température ambiante en moins d’une heure), ce qui est cohérent avec la plus grande réactivité de ce dernier.
J’ai pu confirmer cette coordination par une expérience simple de spectroscopie UV-visible, où l’on peut observer un décalage vers le rouge de la bande MLCT en présence de ligand (Figure 1.10D). La substitution d’un atome attracteur d’électrons (le chlore) par un donneur neutre (le doublet non liant d’azote d’une base de l’ADN) augmente la densité électronique du centre métallique et, par conséquent, diminue l’énergie de la MLCT [71]. Je découvre à cette occasion mon appétence pour la caractérisation d’acides nucléiques par des techniques spectroscopiques “simples”, ce que j’aurai l’opportunité d’exploiter à plusieurs reprises pendant ma carrière.
Finalement, les complexes [Pd(ttpy)] et [Pd(tMebip)] sont également plus efficaces que leurs homologues à base de platine pour inhiber la croissance des cellules cancéreuses in vitro. Plus généralement, ces résultats démontrent que l’affinité pour l’ADN G4 et le mode d’interaction des complexes métalliques peuvent être modulés en modifiant soit le métal, soit le ligand organique.
À titre personnel, la collaboration avec Valérie et Frédéric s’avérera déterminante pour la suite de ma carrière ; je les rejoindrai en post-doctorat quelques années plus tard.
1.3.2 Ligands oligohétéroaryles acycliques qui discriminent fortement entre diverses topologies G4 en ciblant les sillons
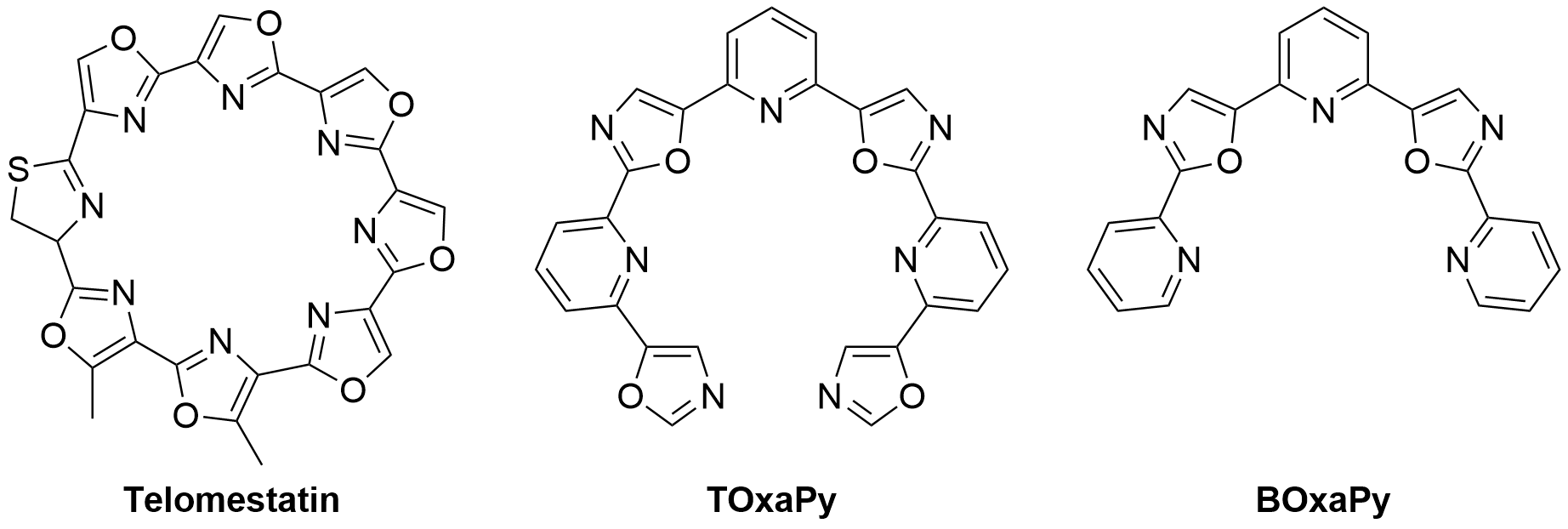
Dans l’optique de potentielles applications thérapeutiques, les ligands de G4 doivent présenter une forte affinité pour le G4 ciblé, combinée à une faible association avec l’ADN duplex. Parmi la pléthore de ligands déjà publiés au début de ma thèse, le premier à avoir répondu à ces critères est la télomestatine, un macrocycle naturel à base d’oxazole (Figure 1.11) [72]. La télomestatine est neutre, ce qui la différencie de la grande majorité des ligands de G4, qui présentent des squelettes aromatiques fusionnés avec des charges cationiques. Sa forme macrocyclique est suspectée d’être le déterminant principal de son interaction avec les G4s et de son absence d’affinité pour l’ADN duplex. Pour répondre à cette question importante dans l’établissement des règles de reconnaissance des G4s, nous avons synthétisé de nouveaux ligands neutres, mais non macrocycliques, à base de motifs oxazole et pyridine alternés (Figure 3A) [73, 74].
Les premiers exemples, TOxaPy et BOxaPy, ont été initiés lors de la thèse de Myriam Rouchon Dagois, soutenue en 2008, sous la direction de Marie-Paule Teulade-Fichou et David Grierson et supervisée par Chi Hung Nguyen.
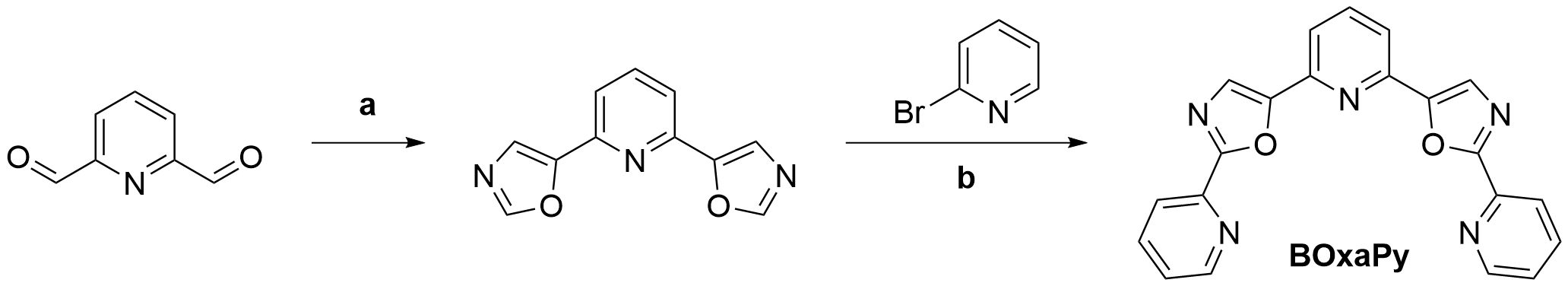
Leur synthèse initiale s’est appuyée sur une procédure convergente basée sur le couplage de la 2,6-bis(oxazol-5-yl)pyridine avec des dérivés de la 2-bromopyridine par la double activation des CH des oxazoles [75] (Figure 1.12). La 2,6-bis(oxazol-5-yl)pyridine a été obtenue en deux étapes par l’oxydation de la 2,6-lutidine avec du dioxyde de sélénium pour obtenir la pyridine-2,6-dicarbaldéhyde [76], qui a été soumise aux conditions de Van Leusen. Le couplage dans des conditions classiques avec deux équivalents molaires de 2-bromopyridine a conduit au penta-hétéroaryle BOxaPy (bisoxazoletrispyridine). La même réaction de couplage a été effectuée en utilisant le 2-bromo-5-(pyridine-2-yl)oxazole pour préparer l’homologue heptaaryle TOxaPy (tétraoxazoletrispyridine).
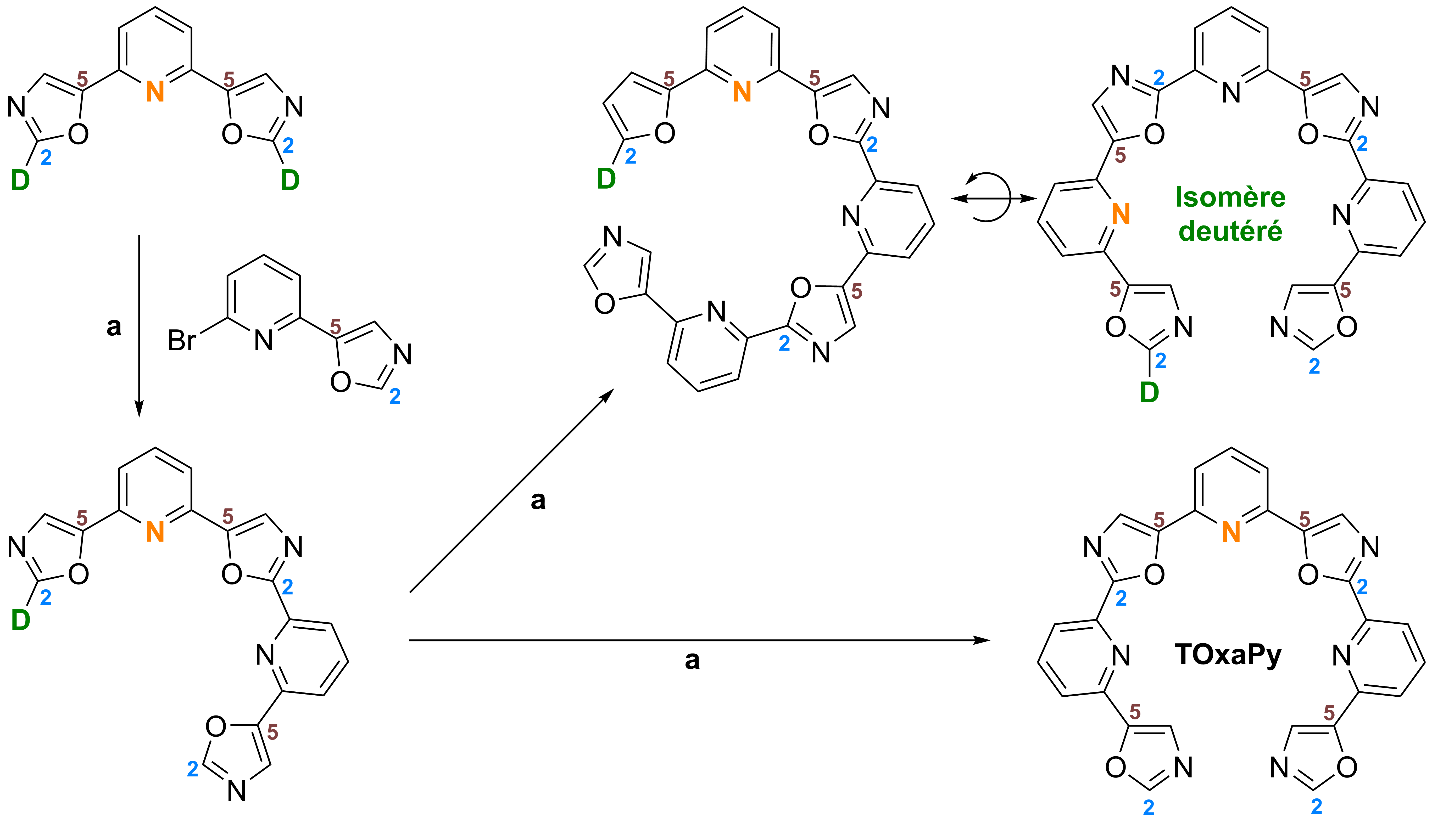
Nous nous satisfaisons de l’analyse en masse qui confirme l’obtention de TOxaPy. Cependant, un reviewer remarque, fort justement, qu’après la première condensation de 2-bromo-5-(pyridine-2-yl)oxazole, la seconde peut avoir lieu soit sur l’oxazole symétriquement opposé au premier site de condensation, soit sur l’oxazole qui vient d’être introduit (Figure 1.13). Cela produit un isomère géométrique de même masse, ne différant que par une jonction pyridine-oxazole. Nous avons confirmé la clairvoyance du reviewer par analyse LC et MS à partir de la 2,6-bis(oxazol-5-yl)pyridine deutérée sur les positions 2 des oxazoles : il y avait bien deux isomères en proportions 50/50 !
Il s’agit d’un moment clé de ma thèse, où l’importance de l’utilisation de techniques analytiques multiples et orthogonales, de la révision par les pairs, et de la remise en question permanente des résultats deviennent évidentes. Je ne le sais pas encore, mais ce premier marquage isotopique au deutérium en appellera beaucoup d’autres.
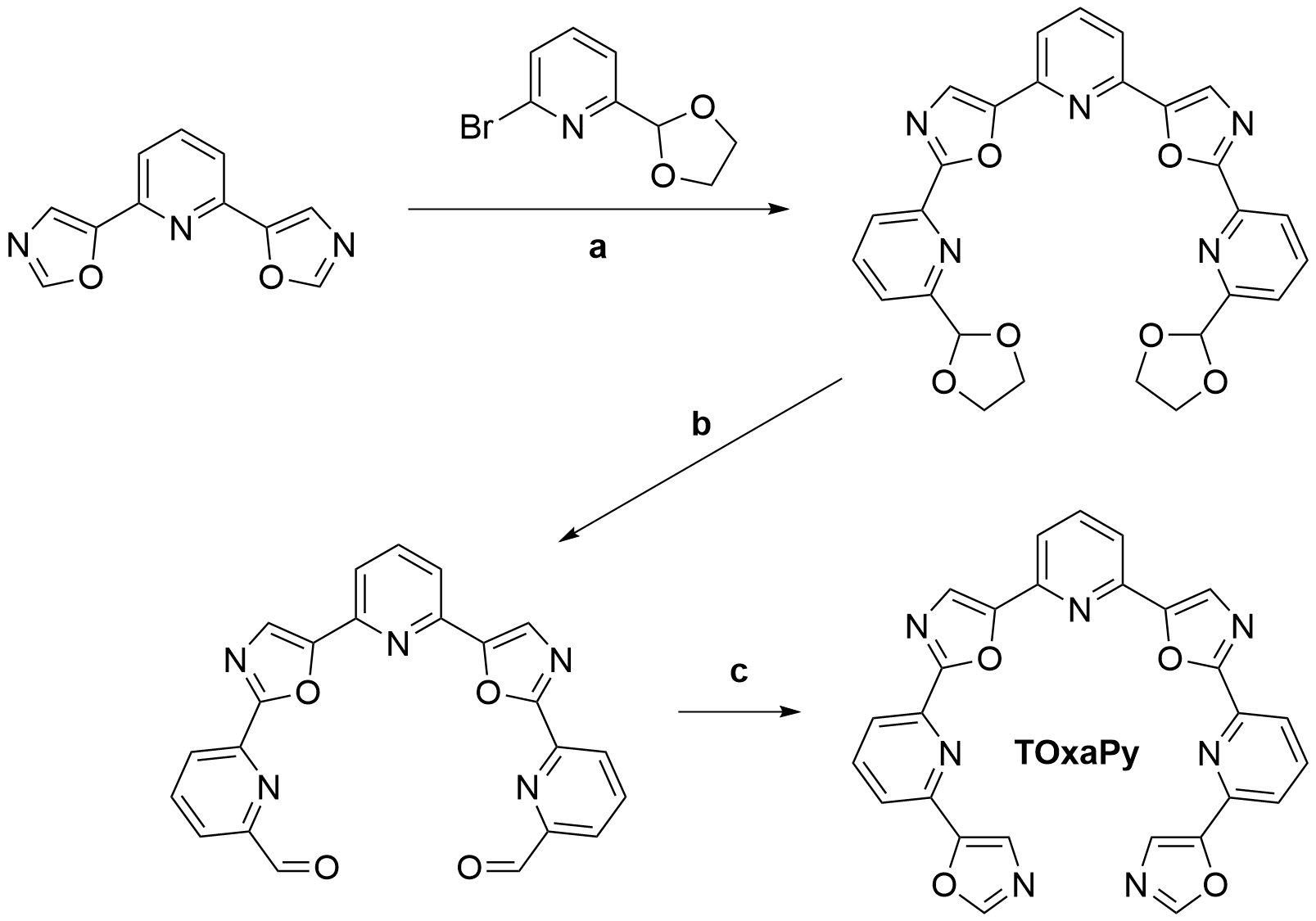
Pour obtenir le TOxaPy sans ambiguïté, Florian Hamon et moi avons suivi une nouvelle voie dans laquelle des groupements oxazoles terminaux ont été synthétisés à partir d’intermédiaire bisformyle pentacyclique correspondants (Figure 1.14). Dans cette approche, la 2,6-bis(oxazol-5-yl)pyridine a été couplé à la 2-bromo-6-(1,3-dioxolan-2-yl)pyridine pour obtenir le précurseur bisacétal qui, après déprotection puis traitement au TosMIC (isocyanure de p-toluènesulfonylméthyl) à produit TOxaPy avec un rendement global de 19%.
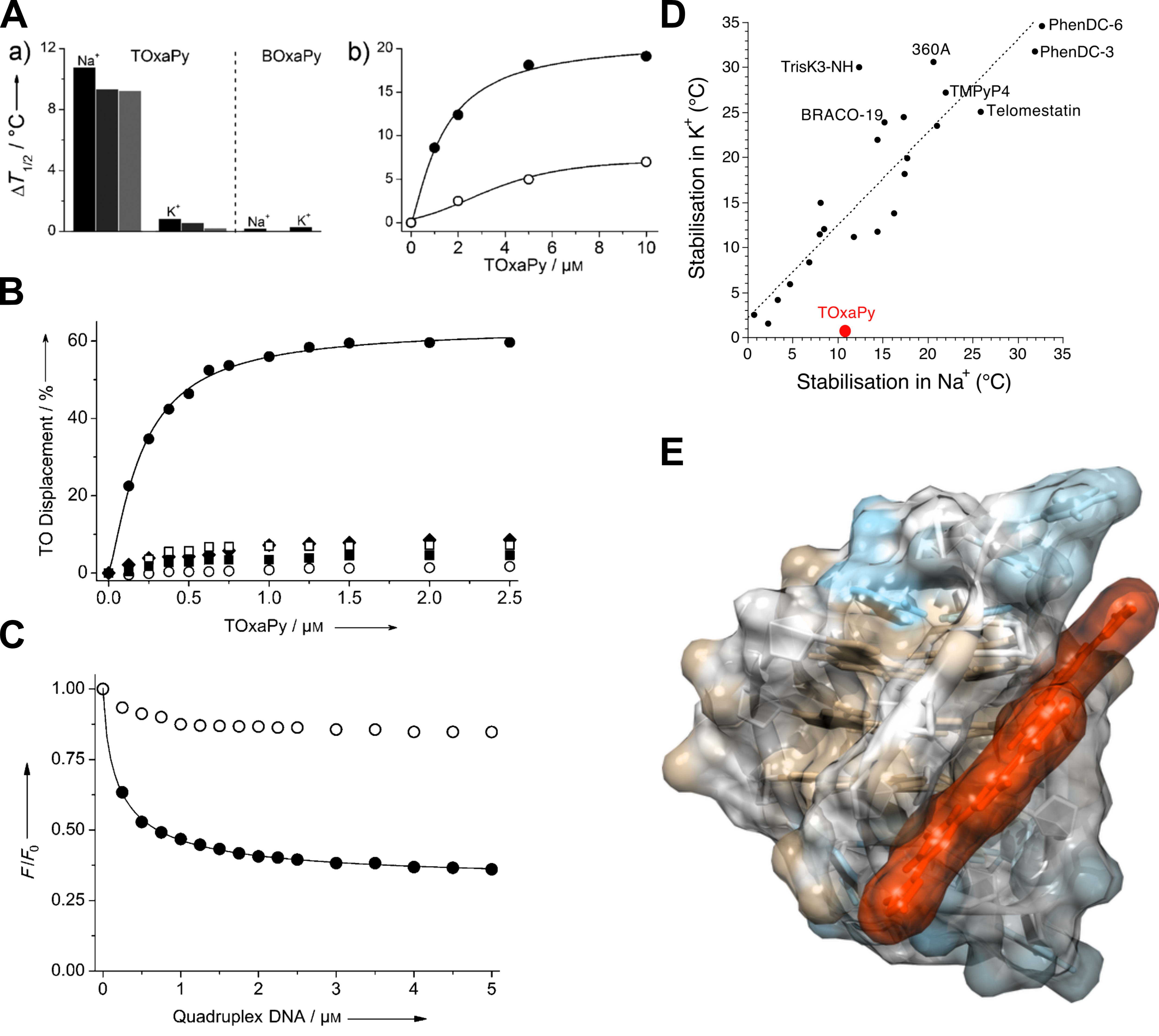
Le dérivé heptacyclique TOxaPy s’est révélé totalement dépourvu d’affinité pour l’ADN duplex tout en présentant une préférence sans précédent pour certaines topologies de G4 par rapport à d’autres. En particulier, le nouveau composé reconnaît la topologie G4 (antiparallèle) de la séquence télomérique humaine formée un tampon riche en Na+, mais pas celle obtenue dans un tampon riche en K+ (mélange de topologies hybrides) (Figure 1.15A). Pour confirmer les résultats du classique FRET-melting (Aurore Guédin et Jean-Louis Mergny, ARNA), j’ai utilisé le test G4-FID (voir Section 1.4.1) et des titrations fluorimétriques - TOxaPy étant fluorescent à l’état libre (\(\lambda_{exc}\): 340 nm, \(\lambda_{em}\): 350-550 nm) et quenché lorsque complexé (Figure 1.15B-C). Cette sélectivité est unique parmi les ligands publiés jusque-là (Figure 1.15D). L’interaction dépend également fortement de la taille de l’oligomère, car l’analogue pentacyclique BOxaPy n’interagit pas avec les G4s (Figure 1.15A).
On notera que, contrairement à la télomestatine, TOxaPy n’est pas un macrocyle. Malgré cela, il est présenté dans la publication (et ici) sous forme circulaire, sans doute pour ressembler à une télomestatine à laquelle il manquerait un hétérocycle. Cela induit un biais de communication visuel important, puisqu’il suggère au lecteur que ces molécules interagissent avec les G4s de façon similaire. Or, les expériences de G4-FID (Figure 1.15B), qui confirment l’absence d’interaction de TOxaPy avec les G4s télomériques en solution K+, suggèrent que la sélectivité de ce ligand s’explique par son interaction dans les sillons de G4, plutôt que par \(\pi\)-stacking sur les G-quartets. En effet, TOxaPy ne déplace que partiellement le thiazole orange, ce qui est cohérent avec une interaction n’ayant pas lieu sur les tétrades. L’interaction dans les sillons est possible, comme mis en évidence par docking par Nicolas Saettel, alors maître de conférences dans l’équipe, sans que cela ne présente une preuve formelle (Figure 1.15E).
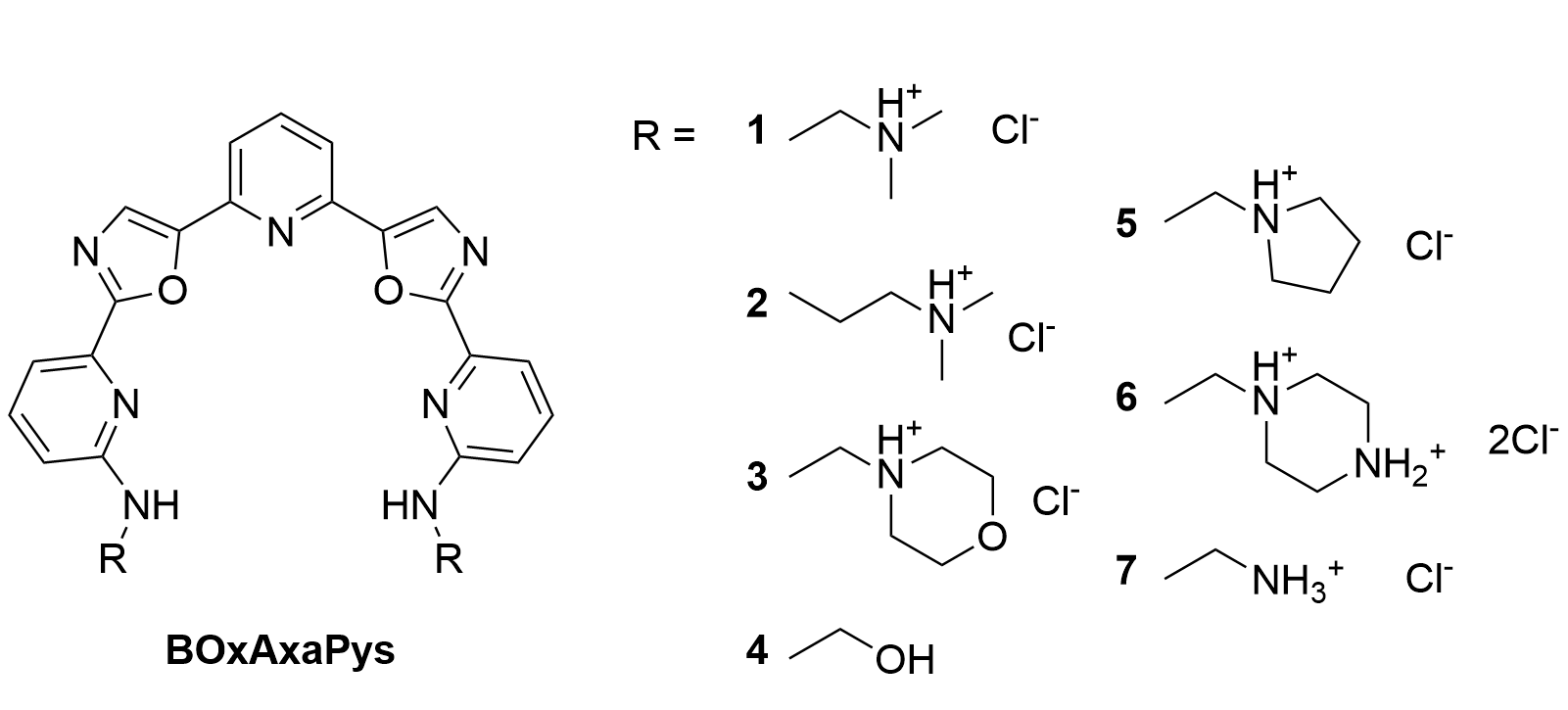
Le principal problème de TOxaPy pour des études in vitro et in vivo est sa très faible solubilité dans l’eau. Une seconde série de dérivés hétérocycliques non condensés “BOxAzaPys”, solubles dans l’eau, a ensuite été préparée en collaboration avec l’équipe du Prof. M. Freccero (Pavie, Italie) (Figure 1.16) [74]. De façon très intéressante, ces composés BOxAzaPys ont une sélectivité inversée, probablement également due à une interaction dans certains sillons (Figure 1.17). Les chaines aminées apportent d’une part la solubilité dans l’eau, et d’autre part modulent l’affinité et la sélectivité de ces composés.
Les sillons de G4s pourraient présenter des géométries plus « uniques » que ne le sont les G-quartets, et seraient donc des cibles à explorer pour obtenir des ligands sélectifs d’une topologie particulière. Ce mode d’interaction est connu pour les duplexes (et permet d’ailleurs d’améliorer la sélectivité des ligands pour des séquences particulières par rapport aux intercalants), mais n’avait pas encore été mis en évidence pour les G4s et reste à ce jour très rare [1]. Notons que, si tous les G-quartets sont similaires, ils présentent un environnement qui peut varier ; nous avons récemment exploré cette opportunité de discrimination (Section 5.3) [59].
1.3.3 Ligands de G4 possédant des propriétés physico-chimiques d’intérêt
Comme indiqué ci-avant, une partie de mes travaux de thèse était liées à la synthèse et caractérisation de ligands de G4s possédant des fonctionnalités d’intérêt. Deux stratégies ont été employées : i. la fonctionnalisation de ligands connus (Section 1.3.3.1) et ii. la synthèse de ligands originaux (Section 1.3.3.2).
1.3.3.1 Dérivés fonctionnalisés de pyridodicarboxamides
Les ligands pyridodicarboxamides (« PDC »), comme 360A (Figure 1.6), sont parmi les plus affins et sélectifs pour l’ADN G4. J’ai été impliqué dans plusieurs projets visant à leur joindre des propriétés d’intérêt. Ainsi, nous avons synthétisé les ligands suivants :
un dérivé biotinylé qui a permis la conduite d’une expérience de SELEX pour la découverte d’aptamères G-quadruplex [77],
un dérivé fluorescent, comme sonde d’ADN quadruplex [78],
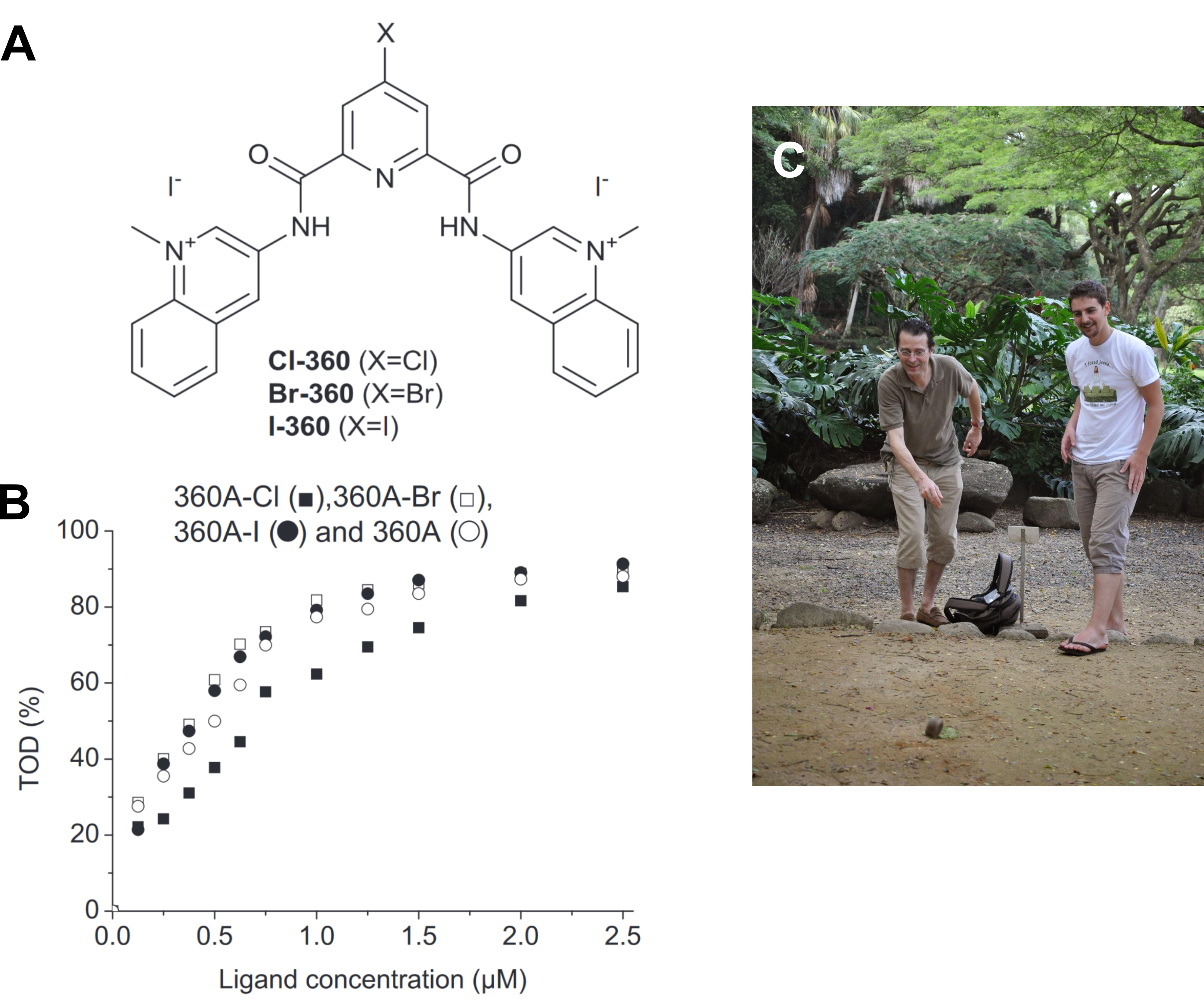
Des dérivés halogénés ont été synthétisés pour leur plus grande affinité et effets aux télomères [79].
Ces ligands sont décrits plus en détails en Sections 1.4.1.3.4 et 1.4.1.3.5, dans le contexte de l’utilisation de leurs propriétés.
1.3.3.2 Ligands fluorescents distyryles
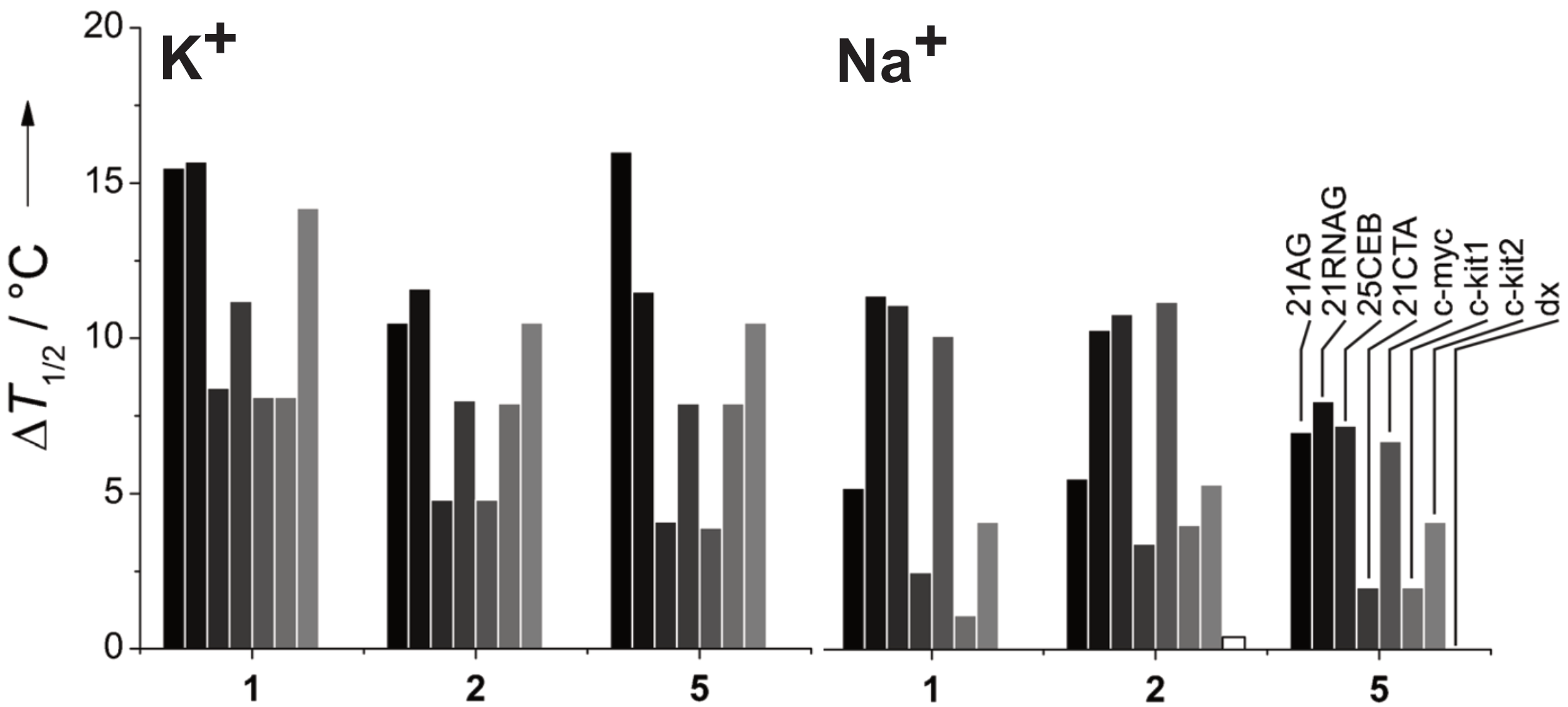
En plus du dérivé fluorescent de PDC, une nouvelle famille de composés distyryles asymétriques fluorescents a été préparée par Xiao Xie, un doctorant (désormais ingénieur R&D chez Technology Transfer Acceleration Company / Institut Pasteur) supervisé par Anton Granzhan, et étudiée de façon à mieux comprendre comment des différences structurales subtiles influencent à la fois leurs fluorescence et sélectivité structurale (Figure 1.18) [80].
Ces dérivés interagissent sélectivement avec l’ADN G4 et ont une forte émission de fluorescence dans le rouge lorsqu’elles sont complexées (sonde fluorescentes off/on). Leur sélectivité optique, ils sont plus de 100 fois plus fluorescent en présence de G4 qu’en absence d’ADN, ou en présence d’ADN duplex, a été mise en lumière par des titrations fluorimétriques.
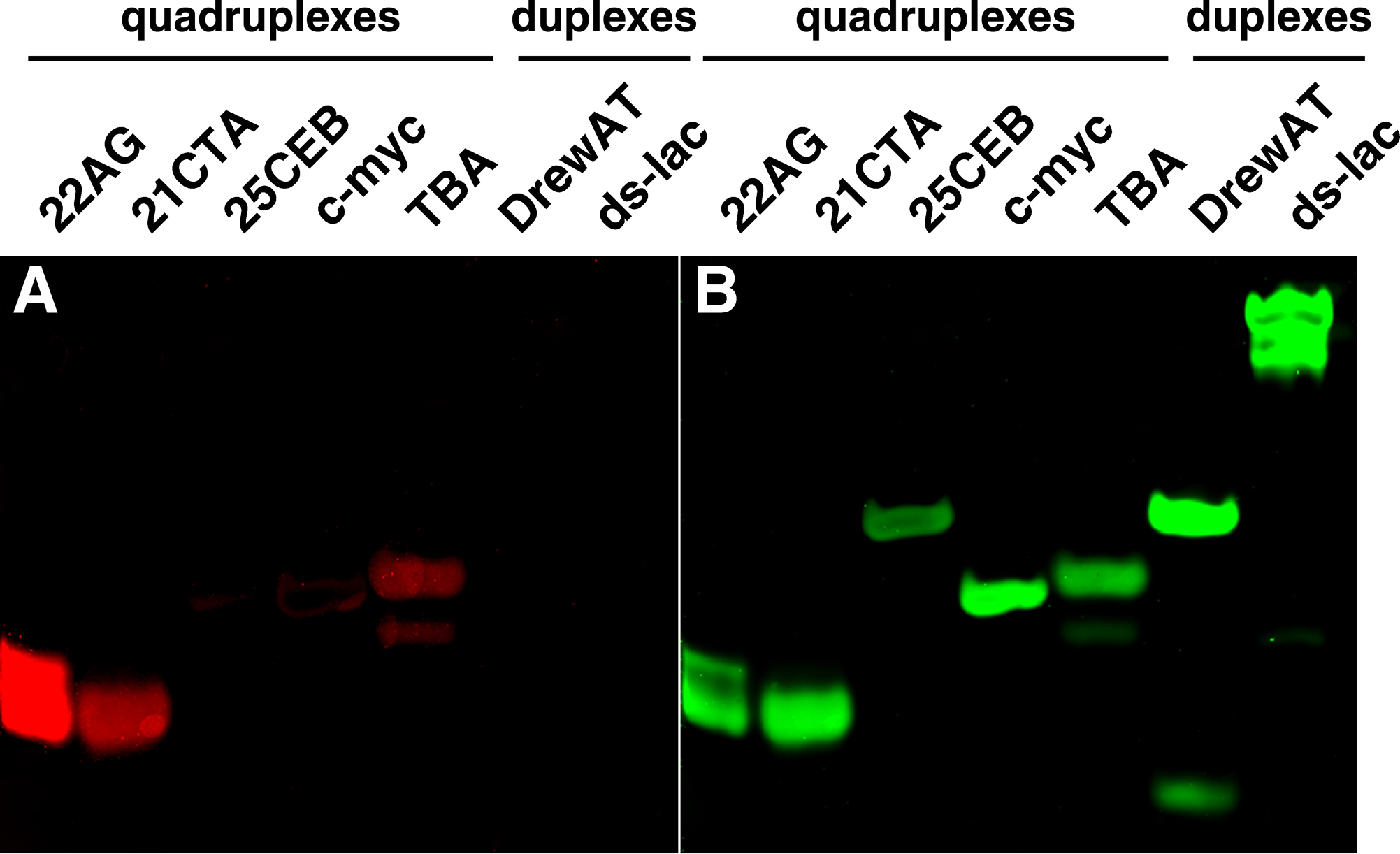
Lors de précédents travaux, j’avais observé que l’ADN G4 était mal visualisé dans des gels natifs par des colorants fluorescents courants tels que le bromure d’éthidium, GelRed ou SYBR Safe, ce qui avait également déjà été noté dans des publications d’autres laboratoires [81–83]. Nous avons observé que la coloration des gels PAGE avec des solutions de 1a ou 1b (2 mM), suivie d’une détection de la fluorescence à 670 nm, permet de détecter sélectivement les bandes correspondant au quadruplexe télomérique (22AG) et à c-myc (Figure 1.19).
En revanche, presque aucune fluorescence n’a été observée dans les bandes contenant des ADN duplex (ds-lac, DrewAT) et TBA. Ce quadruplexe à deux tétrades est très souvent peu lié par les ligands de G4, comme j’aurai l’occasion de le voir dans de nombreuses autres études présentées ici.
Mes contributions à la caractérisation de ces composés fluorescents, à la synthèse de complexes de platine fluorescents (jamais publié ; Figure 1.20) et l’utilisation du plusieurs autre sondes lors de mes travaux de développement analytique (Section 1.4.1), m’ont amené à participer à la rédaction d’une revue exhaustive sur le sujet [84].
1.4 Axe 2. Méthodes analytiques pour l’analyse d’interactions ligands·acides nucléiques
1.4.1 Déplacement de sondes fluorescentes de G4 a haut débit: le test HT-G4-FID
1.4.1.1 Développement du HT-G4-FID
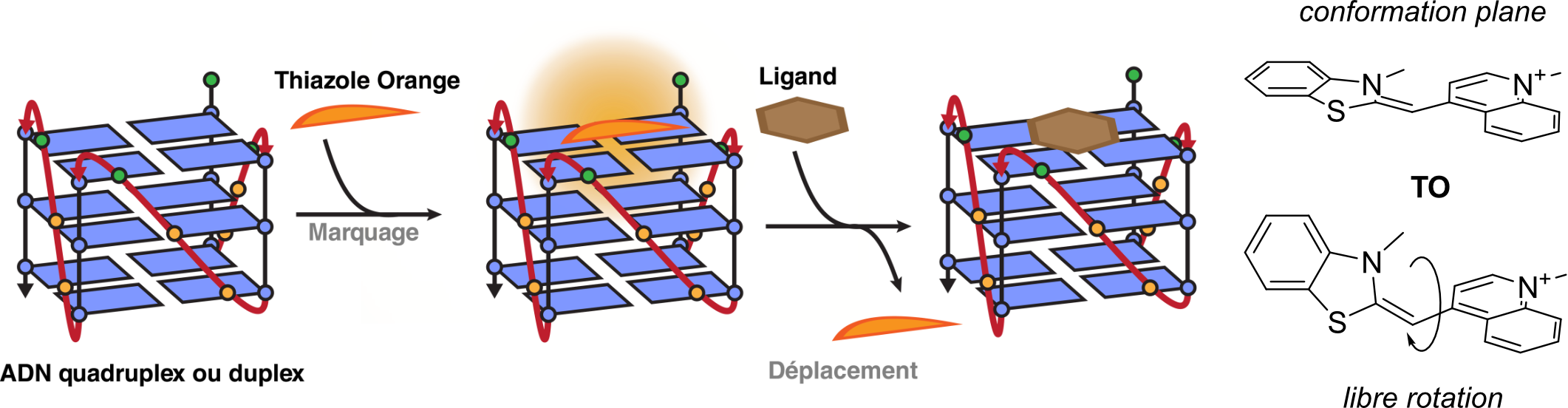
Le FID (Fluorescence Intercalator Displacement) est une technique qui permet de déterminer l’affinité de molécules a priori non fluorescentes (et ne possédant pas un autre type de propriété physico-chimiques permettant une titration) pour des oligonucléotides de diverses structures (quadruplexes, duplexes, hairpin, triplex), par le déplacement d’une sonde fluorescente off/on (Figure 1.21). Cette technique a été développée par Dale L. Boger (Scripps Research Institute), que j’ai eu la chance de voir présenter lors du Pacifichem 2010 [85–88].
Concrètement, l’oligonucléotide est marqué de façon non covalente par une sonde dont la fluorescence est fortement exaltée en sa présence (on ; structure plane et isolé du solvant aqueux). L’ajout de quantités croissantes de ligand entraîne le déplacement de la sonde et donc une diminution de la fluorescence (off ; structure en libre rotation et accessible par l’eau; Figure 1.21). Le suivi de l’intensité de fluorescence permet d’estimer l’affinité de la molécule pour l’oligonucléotide testé. Plus un ligand est affin, plus de faibles concentrations de celui-ci suffisent à déplacer la sonde.
Pour son application aux structures G-quadruplexes, on parle de test G4-FID et la sonde choisie par David Monchaud (parti depuis à l’Université de Bourgogne) lors du développement initial de la méthode est le thiazole orange (TO ; Figure 1.21), plutôt que le bromure d’éthidium (EtBr), qui lui est préféré pour l’ADN duplex, mais fluoresce peu avec les G4s. On notera que :
Dale Boger, qui utilisait principalement le EtBr, avait testé le TO comme sonde pour du FID sur des tiges-boucle d’ADN, en concluant qu’il constitue une alternative dans les cas où l’excitation ou l’émission des ligands étudiés chevauche celle du EtBr [86]. C’est dans cette même optique que j’explorerais l’utilisation de sondes alternatives,
Le FID porte mal son nom quand appliqué aux G4s car le TO ne s’y intercale pas.
Les avantages du FID sont nombreux. C’est une méthode peu chère, rapide (notamment en microplaque ; voir les sections 1.4.1.2.1, 1.4.1.2.2), exacte et précise, et dotée d’une bonne résolution. Par rapport au FRET-melting, elle a l’avantage d’être isotherme et ne nécessite qu’un fluorimètre standard. En revanche, elle ne permet généralement pas de mesurer de constantes d’affinité, même si cela est possible dans certains cas particulier que j’ai eu la chance de rencontrer (Section 1.4.1.3.2). Le FID peut aussi apporter des informations qualitatives sur le mode d’interaction. Par exemple, le déplacement partiel du TO par le ligand TOxaPy suggère que ce dernier n’interagit pas sur les G-quartets, comme le TO, mais plutôt dans les sillons (Figure 1.15) [73].
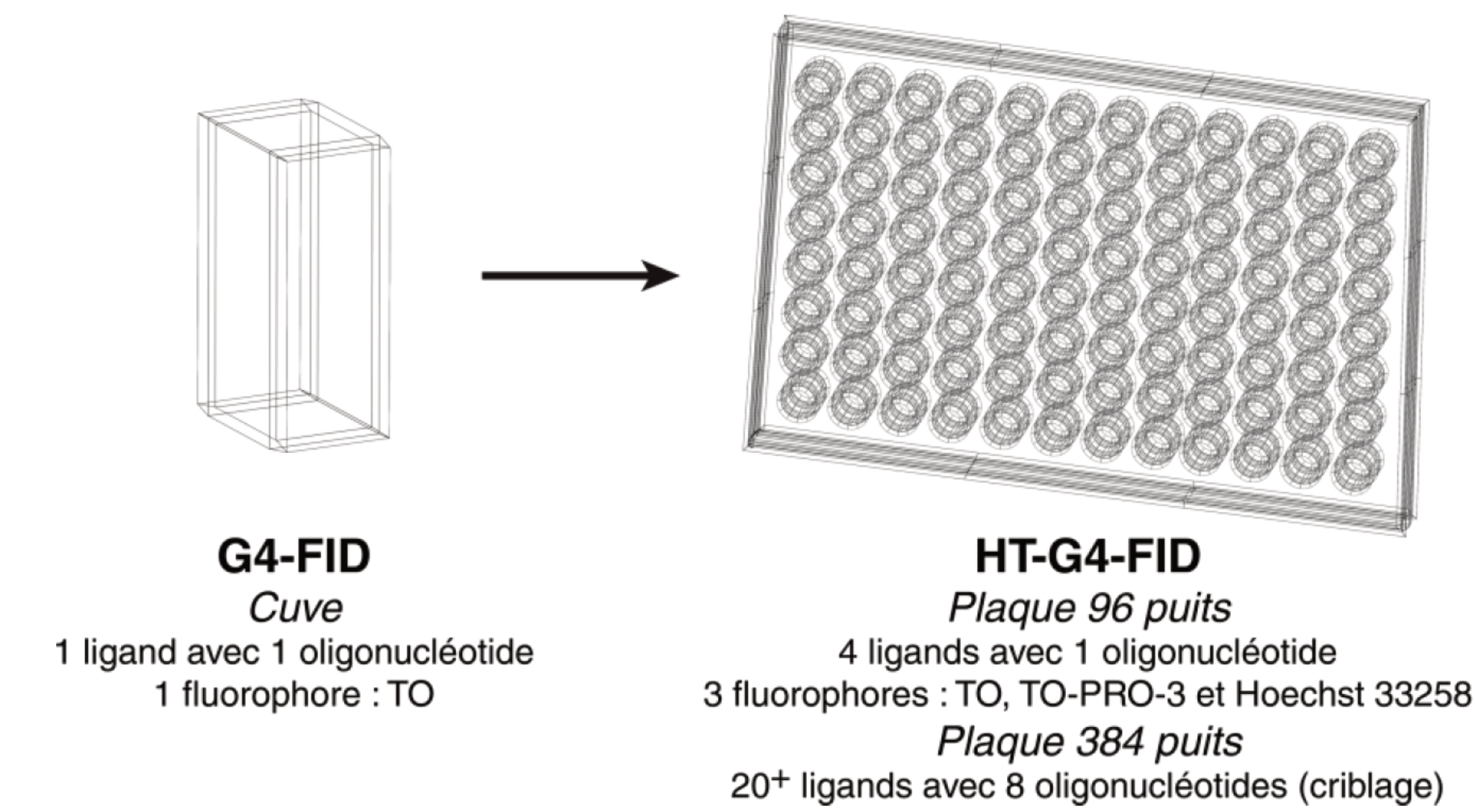
Après avoir utilisé la méthode telle que mise au point par David Monchaud (avec le TO), ou dans la version de Dale Boger (avec l’EtBr ; Section 1.4.1.3.1), en cuvettes, j’ai souhaité développé une version à haut débit de l’essai, nommée HT-G4-FID (High-Throughput Quadruplex Fluorescent Intercalator Displacement), qui est réalisé dans des microplaques à 96 ou 384 puits (Figure 1.22) [89, 90]. En effet, la réalisation du test en cuve implique une acquisition point par point des titrations, avec agitation manuelle de la cuve, et cela pour chaque couple G4/ligand. L’utilisation de plaques permet de réaliser plusieurs titrations entières en parallèle, et ouvre la porte à l’automatisation des opérations de pipetage, ce que j’ai mis en œuvre ultérieurement (Section 1.4.1.2.2).
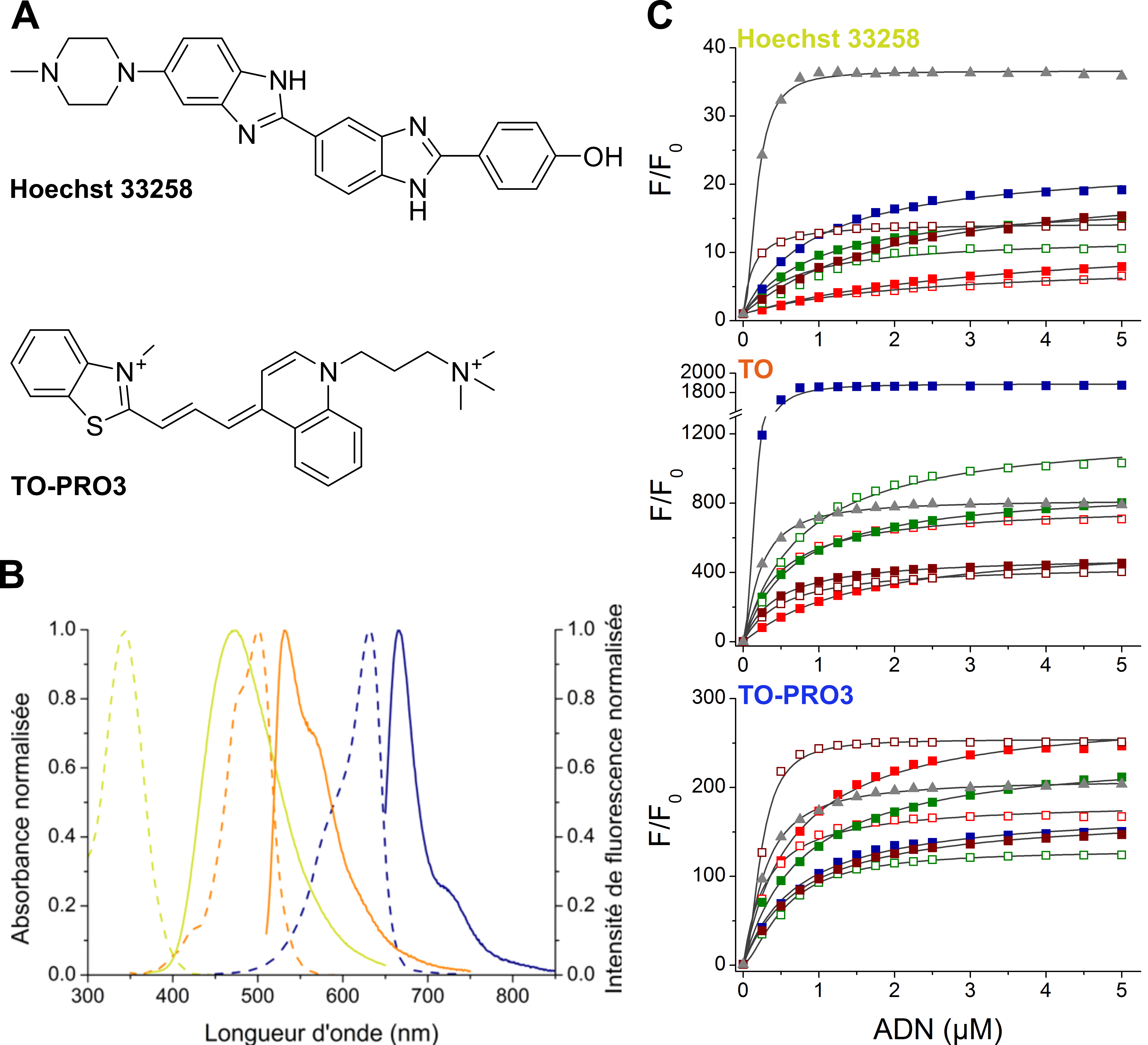
Dans le même temps, et à l’instar de D. Boger, j’ai étendu la gamme spectrale de l’essai en utilisant deux sondes fluorescentes supplémentaires (Hoechst 33258 et TO-PRO3 ; Figure 1.23), dont les spectres d’absorption et d’émission sont complémentaires de ceux du TO [89]. Cela permet de cribler des ligands susceptibles d’absorber et/ou d’être fluorescents dans la même gamme spectrale que le TO (comme la porphyrine TMPyP4), et donc de fausser les résultats. Ces améliorations ont été validées avec un panel cinq oligonucléotides d’ADN et cinq ligands d’affinité et sélectivités variées.
Le test HT-G4-FID peut donc être appliqué à haut débit pour n’importe quel couple petite molécule / séquences d’ADN ou d’ARN de structures diverses sans qu’aucune modification soit nécessaire. Il constitue donc un outil puissant pour la découverte de nouveaux ligands capables de se lier sélectivement à l’ADN G4 vs. l’ADN duplex mais aussi entre différents G4s. Le test HT-G4-FID est fréquemment employé par la communauté scientifique, comme en témoigne le nombre élevé de citations (plus de 280 au total) [89, 90]. Je présente ci-dessous mes applications les plus significatives de ce test, et par la même occasion certains projets auxquels j’ai pu contribuer pendant ma thèse.
Pour le développement de cette méthode, et certaines applications j’ai encadré deux étudiants : Camille Valette et Yoann Lebreton (BTS, École nationale de chimie physique et biologie de Paris).
1.4.1.2 Découverte de ligands sélectifs de G4s par criblage
1.4.1.2.1 Exploration d’une ciblothèque
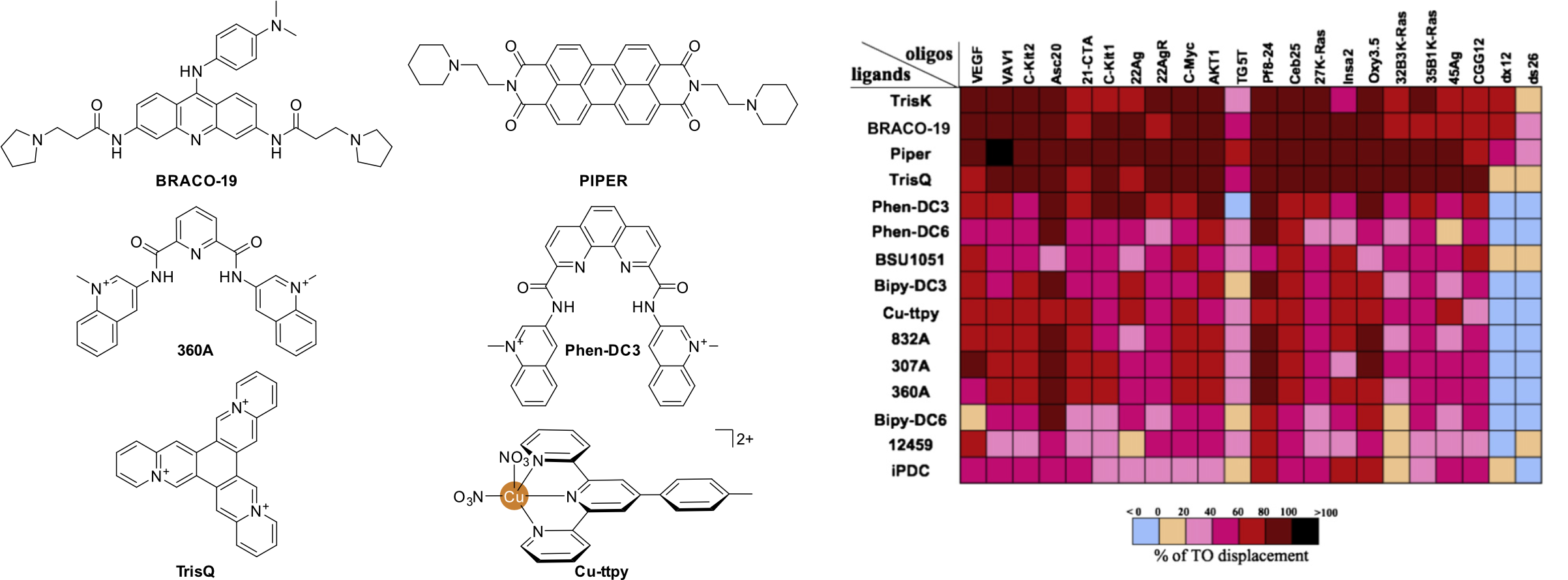
Dans le cadre du développement du HT-G4-FID, j’ai collaboré avec l’équipe de J.-L. Mergny (Muséum National d’Histoire Naturelle, Paris) pour cribler quinze ligands de G4s connus appartenant à différentes familles contre vingt G4s et deux duplexes contrôle [90]. Nous avons noté de très larges différences d’intensité de fluorescence du TO en fonction des oligonucléotides, ce que nous avions déjà observé précédemment mais dans une moindre mesure (Figure 1.23), et éliminé ceux pour lesquelles la fluorescence ne permettait pas de réaliser le test avec une grande sensibilité.
La plupart des composés a montré une bonne sélectivité en faveur des G4s contre les duplexes (Figure 1.24). En revanche, ils ont peu ou pas de spécificité pour une séquence G4 par rapport aux autres, ce qui limite leur potentiel thérapeutique.
1.4.1.2.2 Exploration d’une chimiothèque
J’ai présenté ci-avant la synthèse de ligands présentant une sélectivité pour certaines topologies G4s [73, 74]. Si cette avancée était significative, elle n’était pas le produit d’un design rationnel. Il n’existait pas, et n’existe toujours pas, de « guide moléculaire » décrivant les règles à suivre pour cibler une structure en particulier. J’ai alors estimé que le design rationnel ne pouvait pas être la seule approche pour obtenir des ligands de G4s présentant des sélectivités significatives pour certains G4s.3
3 Ce qui m’arrangeait bien étant donnée ma faible appétence pour la synthèse organique.
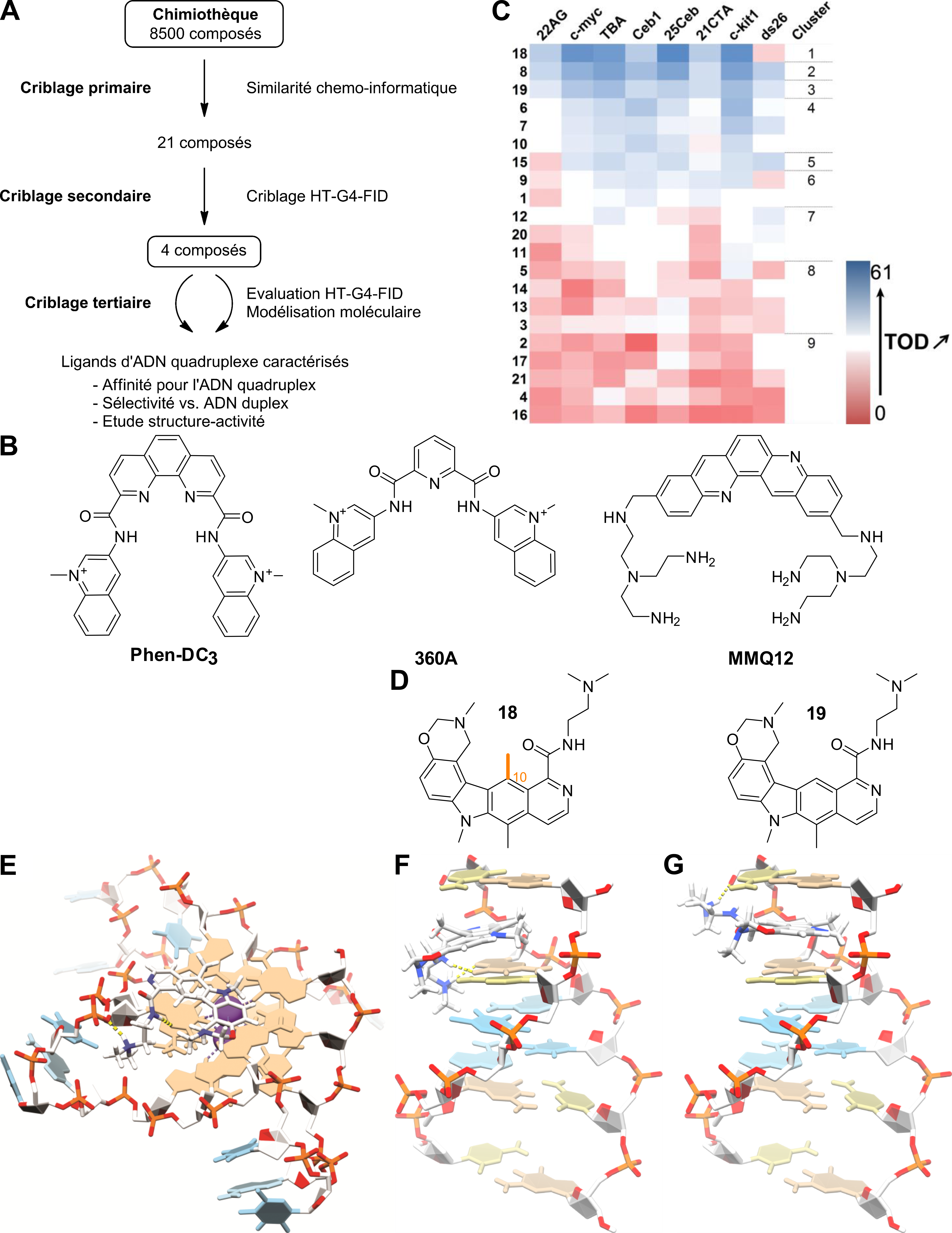
En tant qu’approche complémentaire, le criblage peut fournir de nouvelles pistes de travail pour peu que des outils analytiques adaptés soient disponibles [91], ici le test HT-G4-FID. J’ai criblé la chimiothèque de l’Institut Curie/CNRS pour identifier des candidats, avec l’assistance de deux élèves ingénieures, Valentine Boyer (EBI, Cergy) et Marie Perrot (ESCOM, Cergy) (Figure 1.25A) [92].
La première étape a permis la sélection des candidats les plus prometteurs par des méthodes chimio-informatiques, en particulier des calculs de similarité moléculaire avec des ligands de référence (PhenDC3, 360A, MMQ12 ; Figure 1.25B). Les molécules retenues ont été évaluées par HT-G4-FID contre divers G4s et un duplex contrôle (Figure 1.25C). La compréhension de la relation structure/activité de ces quatre ligands ellipticines (Figure 1.25D) s’est également appuyée sur des expériences de docking (Figure 1.25E). Nous avons ainsi mis en évidence que seul le ligand ayant un groupe méthyle en position 10 était sélectif contre l’ADN duplex, car il entraîne une perte de planéité de la molécule, ce qui en fait un mauvais intercalant (Figure 1.25D,F).
L’approche par similarité chimique n’est pas un très bon choix pour découvrir des ligands de G4 sortant du paradigme actuel (ligands hétérocycliques condensés plans), ce qui n’était cela dit pas le but premier de cette étude. Pour cela, il aurait été préférable de cribler l’entièreté de la chimiothèque, ce qui aurait malgré tout sans doute été vain étant donné que celle-ci était fortement biaisée en faveur de petites molécules hétérocycliques planes (souvent des ligands de duplexes et triplexes), à l’image des ellipticines que nous avons sélectionnées.
Cette expérience m’a amené à écrire un chapitre de livre sur le criblage de ligands de G4s [91]. Si j’en concluais qu’il ne fallait pas se reposer sur la chance seule, il semble que cela soit parfois suffisant. Je n’en dit pas plus pour ne pas divulgâcher la Section 5.3.1.1.
1.4.1.3 Caractérisation de ligands issus du design rationnel
1.4.1.3.1 Déterminants structurels de la sélectivité de ligands macrocycliques pour les paires de bases d’ADN mésappariées
Les nucléotides mésappariés sont des paires de nucléotides au sein de l’ADN duplex qui ne respectent pas l’appariement classique des bases (Watson-Crick) [93]. Les mésappariements peuvent se produire naturellement au cours de la réplication ou de la recombinaison homologue, ou être provoqués par des facteurs externes tels que des molécules mutagènes ou des radiations ionisantes [93, 94]. L’efficacité de la réparation enzymatique des mésappariements dépend de la nature de la paire mal appariée, et des déficiences dans la réparation des mésappariements sont associées à de nombreux cancers [93]. L’inhibition de la réparation est étudiée comme un moyen d’améliorer les effets des médicaments anticancéreux. Dans ce contexte, les molécules qui se lient sélectivement aux paires de bases mésappariées de l’ADN pourraient servir de base à de nouveaux agents chimiothérapeutiques.
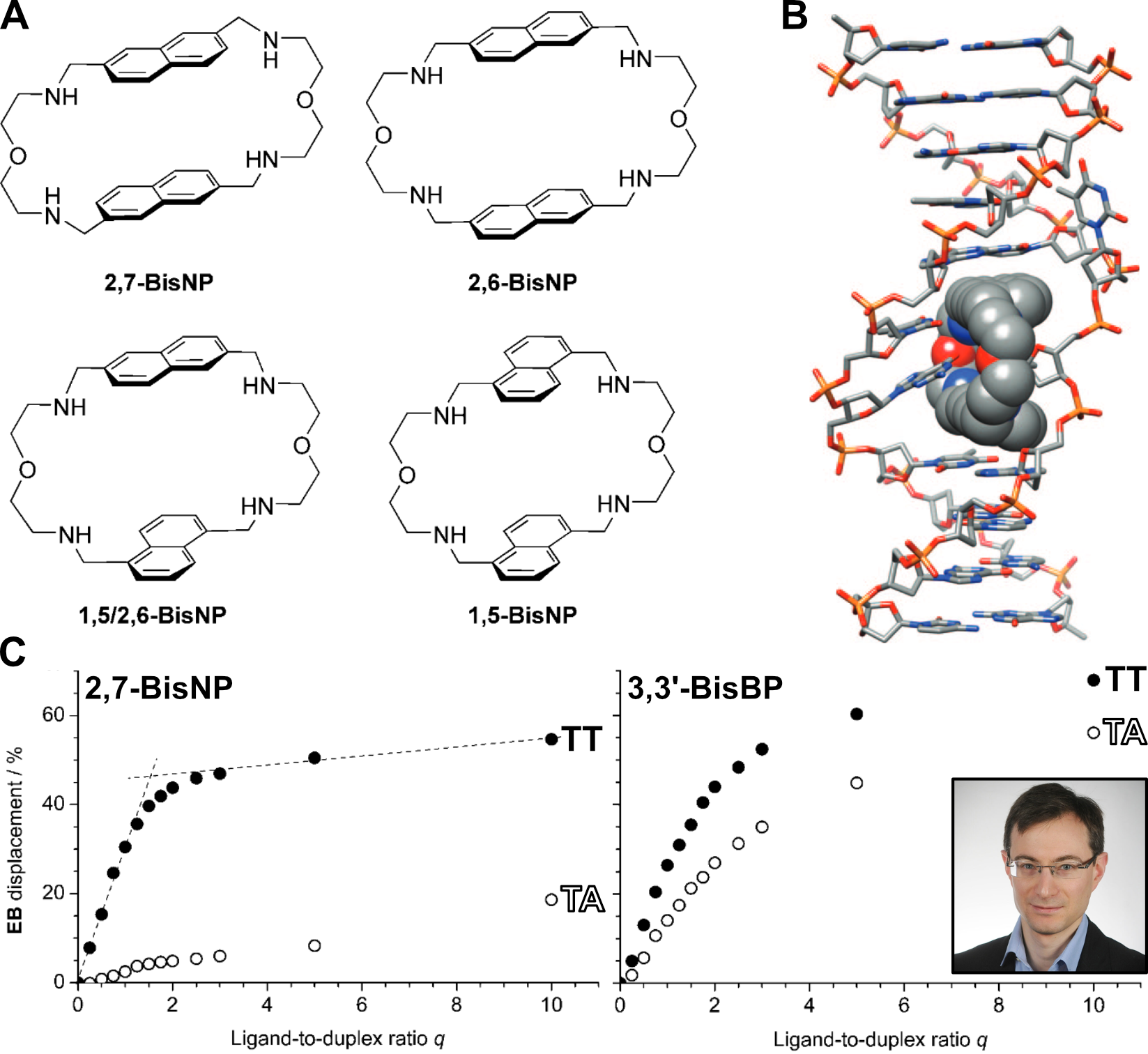
Anton Granzhan avait préparé une série de 20 bis-intercalants macrocycliques lors de son séjour postdoctoral au laboratoire (il y est finalement revenu comme chargé de recherche CNRS, avant de rejoindre l’UMR 8601 de l’Université Paris Cité en 2025) (Figure 1.26A). J’ai été chargé après son départ d’étudier leurs interactions avec des doubles hélices d’ADN, contenant un résidu de thymine mésapparié (TX ; Figure 1.26B) ou correctement apparié (TA), par FID (Figure 1.26C) [95]. L’EtBr a été utilisé comme intercalant plutôt que le TO car il est plus adapté à l’ADN duplex [85].
Les dérivés 2,7-disubstitués du bisnaphtalène ont présenté la plus grande sélectivité pour les mésappariements TX par rapport à l’ADN apparié (Figure 10). Les différences spectaculaires d’affinité et sélectivité entre les diverses séries de macrocycles que nous avons observées soulignent la forte influence de la taille et de la topologie du macrocycle sur la reconnaissance de ces mésappariements. Cette étude a permis d’établir un guide structure-propriété des ligands d’ADN contenant des mésappariements TX.
À titre personnel, il s’agit de ma toute première publication, mais j’ai surtout beaucoup appris de la rigueur scientifique d’Anton, qui reste un modèle pour moi.
1.4.1.3.2 Inhibition de l’expression génique par ciblage des G4s dans les 5’-UTR d’ARN
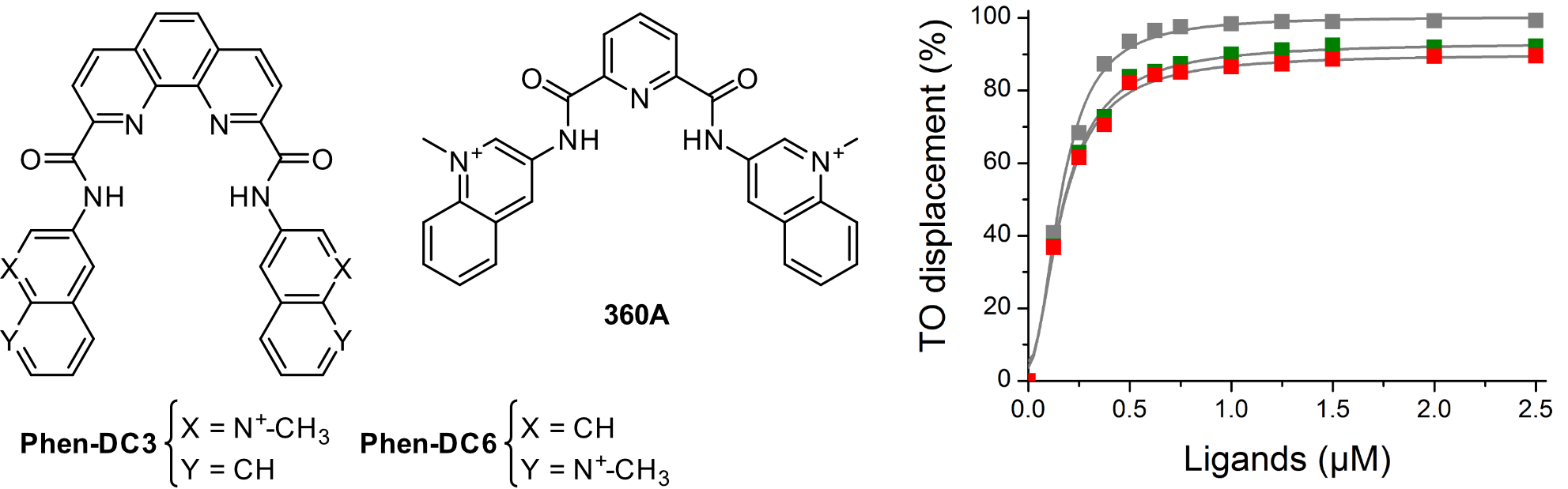
Le Pr. Jörg Hartig (Université de Constance, Allemagne) avait montré que la formation de G4s d’ARN dans Escherichia coli inhibe l’initiation de la traduction car ils interfèrent avec l’accessibilité du site d’interaction des ribosomes [96]. Des effets inhibiteurs similaires ont été démontrés pour les G4s d’ARN naturels basés sur le 5’-UTR (régions non traduites) dans la culture de cellules de mammifères pour l’expression de certains gènes [97–100]. En outre, le niveau de suppression est proportionnel à la stabilité thermodynamique du G4 [101]. Le Pr. Hartig a alors initiée une collaboration avec notre équipe pour évaluer la possibilité de réguler in vivo l’expression de gènes qui possèdent des G4 dans leur 5’-UTR grâce à des ligands stabilisant ces G4s [102].
J’ai utilisé le test HT-G4-FID pour évaluer l’affinité des ligands 360A, PhenDC3 et PhenDC6 pour des G4s d’ARN (Figure 1.27). Lors de la caractérisation des différents complexes ARN/TO par titration fluorimétrique, préliminaire à l’application du test HT-G4-FID (comme dans la Figure 1.23), nous avons remarqué l’affinité très faible du TO pour ces G4, probablement plusieurs ordres de grandeur plus faible que celles des ligands bisquinolinium. Loin d’être un désavantage, cette large différence d’affinité m’a permis de déterminer les excellentes constantes d’affinité (1—10 nM) entre ces ligands et les G4s r(G3U)4 et r(G3U2)4. On peut en effet, dans ces conditions bien particulières, “ignorer” l’équilibre entre le G4, le TO et leurs complexes, et procéder comme s’il s’agissait d’une titration directe \(G4 + ligand \rightleftharpoons G4 \bullet ligand\).
Il a ensuite été démontré que l’effet inhibiteur était plus prononcé sur les ARNm présentant une forte propension à la formation de G4, et que cet effet est amplifié quand le G4 est stabilisé par un ligand. Ces résultats suggèrent donc que ces ligands peuvent être utilisés pour réguler l’expression des gènes en ciblant les structures G4 de 5’-UTR d’ARN.
1.4.1.3.3 Instabilité génomique de minisatellites humains déclenchée par des ligands de G4
Les G4s sont des structures secondaires d’acides nucléiques pour lesquelles de nombreux rôles biologiques ont été proposés mais dont l’existence in vivo a longtemps été difficile à démontrer et le sujet de nombreux débats. Généralement, la première question à une présentation sur un sujet “G4” pouvait se résumer à : “Est-ce que ça existe vraiment vos bidules ?”. Il n’y a plus beaucoup de débat aujourd’hui, grâce notamment à l’utilisation d’anticorps anti-G4 [103–107], mais aussi d’études, comme celle ci-dessous, plus indirecte mais pas moins convaincante à mon humble avis.
4 Comme quoi les G4s mènent à tout.
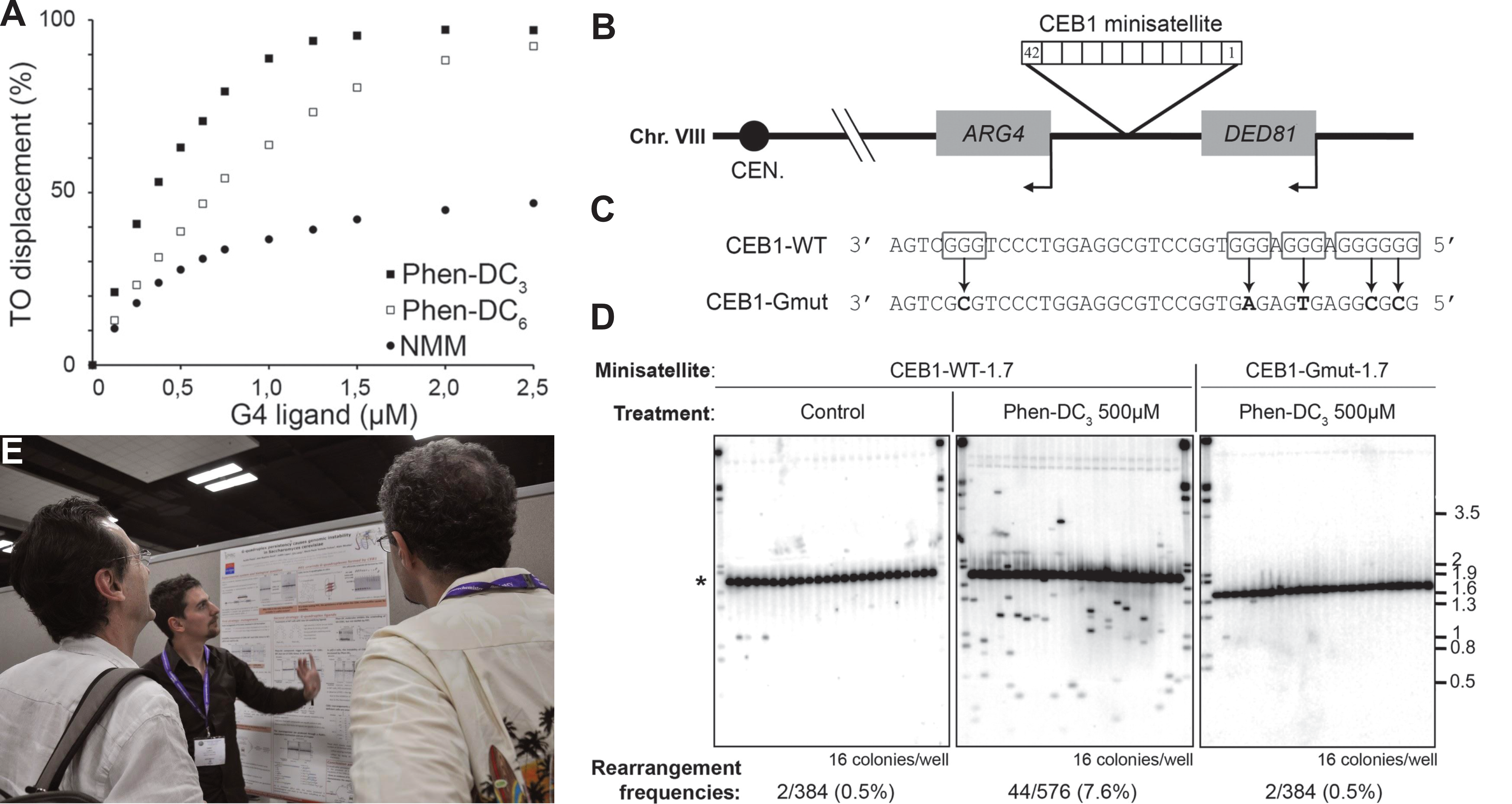
Dans le cadre d’une collaboration avec l’équipe du Dr. Alain Nicolas (Institut Curie, Paris), j’ai ainsi utilisé le test HT-G4-FID pour démontrer la grande l’affinité des ligands PhenDC3 et PhenDC6 pour le G4 formé par le minisatellite humain CEB1, et leur sélectivité par rapport à l’ADN duplex [108]. Cela a permis d’expliquer leur capacité à inhiber la déstructuration de ce G4 par l’hélicase Pif1 chez Saccharomyces cerevisiae. In vivo, les deux composés ont rapidement induit des réarrangements dépendants de la recombinaison de CEB1, mais n’ont pas affecté les séquences ne formant pas de G4. Pour ces expériences, j’ai encadré Katie Mingo, alors étudiante en médecine (MIT, Boston, USA) et désormais oto-rhino-laryngologue a la Cleveland Clinic (Minneapolis, MN, USA).4
1.4.1.3.4 Effets de ligands de G4 halogénés sur la séquence terminale des télomères humains
Des modifications chimiques mineures de structures de ligands de G4 peuvent affecter à la fois leurs propriétés biophysiques et biologiques. En effet, des ligands de structures et d’affinités pour les G4 proches, tels que 360A, PhenDC3 et PhenDC6, ont montré des différences remarquables au niveau des changements transcriptionnels dans les gènes contenant des G4s, ce qui fut démontré dans certaines études ci-dessus et d’autres [90, 102, 109]. D’autre part, l’introduction d’halogènes dans un composé biologiquement actif est souvent utilisée pour améliorer la perméabilité aux membranes (barrières cellulaire et hémato-encéphalique). En outre, les effets stériques et électroniques des atomes d’halogène améliore parfois l’interaction avec des cibles biomoléculaires [110].
Il a été démontré que des ligands de G4 peuvent perturber l’interaction de composants télomériques essentiels, tels que POT1, et déclenchent un dysfonctionnement télomérique associé à un arrêt de croissance retardé dans les cellules tumorales [111–113]. Dans ce contexte, Jean-François Riou a initié une étude sur les effets de ligands de G4s halogénés sur la séquence terminale de télomères de cellules de fibrosarcome HT1080.
J’ai assisté à la synthèse d’une série de dérivés halogénés du ligand 360A (Figure 1.28A), pour laquelle j’ai encadré Julien Botton (Master, Université Pierre et Marie Curie, Paris VI) avec Florian Hamon, puis j’ai évalué leur affinité pour les G4s télomériques humains avec le test HT-G4-FID. Les dérivés bromé et iodé, mais pas le chloré, ont une affinité légèrement supérieure à celle de 360A, tout en conservant une excellente sélectivité contre l’ADN duplex (Figure 1.28B). Malheureusement, nous n’avons pas pu rationaliser le comportement différent du dérivé chloré sur la base de l’électronégativité et de considérations stériques. Au final, les cellules de fibrosarcome HT1080 traitées jusqu’à 24 jours avec 360A-Br ont présenté des variations mineures mais significatives de leur composition en nucléotides terminaux du brin C, avec également une déplétion partielle de POT1.
Comme pour de nombreux sujets explorés au cours de ma thèse, certains aspects de ce travail réapparaitront bien plus tard. Récemment, j’ai ainsi participé à une étude portant sur l’influence de la substitution de ligands phényls de colonnes HPLC sur leurs interactions avec des oligonucléotides (Section 5.6.5) [114].
1.4.1.3.5 Ligands fonctionnalisés pour la sélection d’aptamères anti-G4
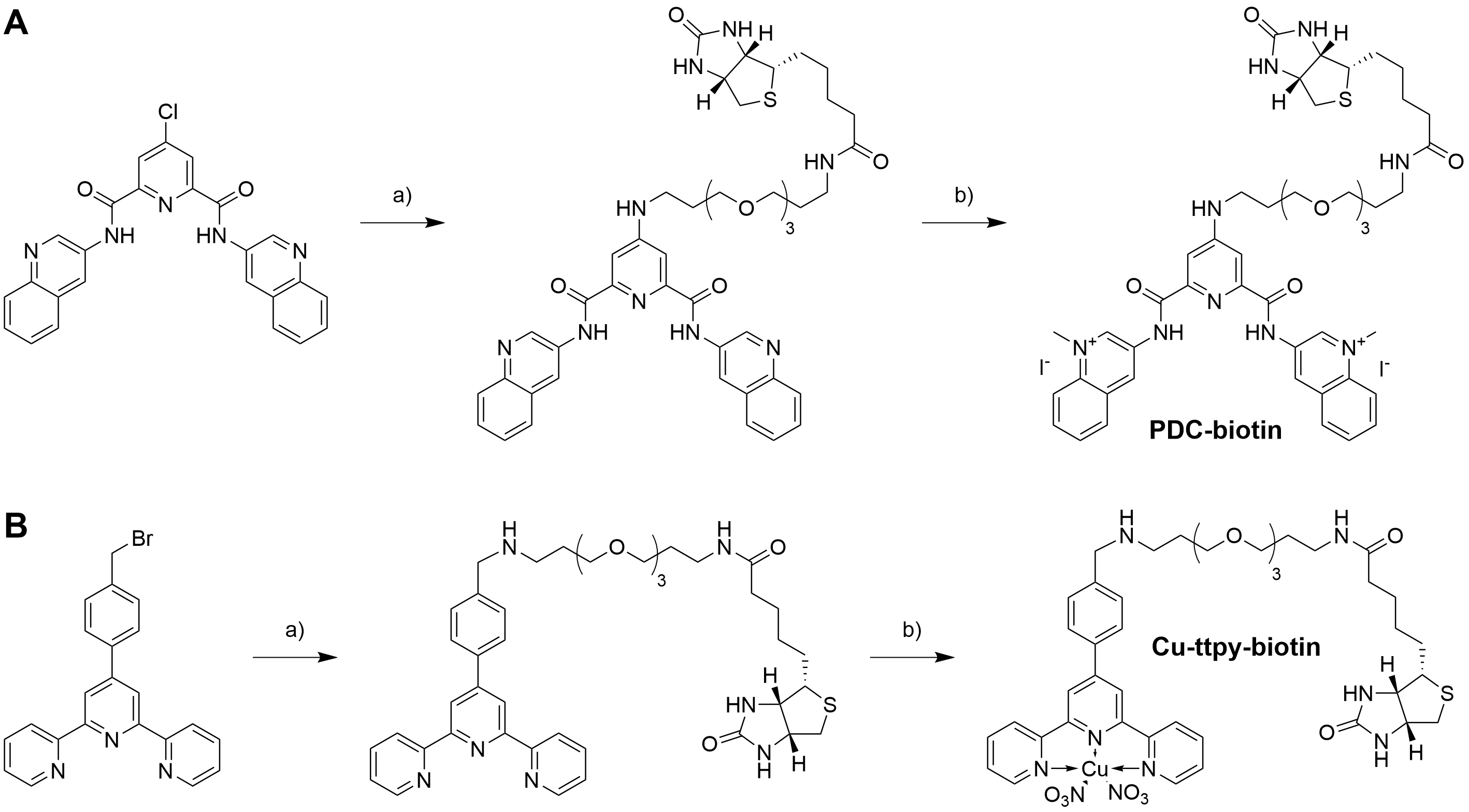
Une collaboration avec les équipes de Jean-Louis Mergny et Jean-Jacques Toulmé (IECB, Bordeaux) avait pour but de sélectionner par SELEX des aptamères d’ADN qui se lient à un ligand de G4 spécifique. Pour cela, il nous fallait fournir des ligands fonctionnalisés avec une biotine de façon à pouvoir les immobiliser lors des expériences de SELEX. Nous avons opté pour deux ligands facilement dérivatisables, 360A (PDC) et Cu-ttpy, auxquels nous avons couplé une biotine avec une chaine suffisamment longue pour permettre leur immobilisation sans compromettre leur affinité pour les G4s.
Florian Hamon est reparti d’un intermédiaire de synthèse de Cl-360A (celui précédant la méthylation finale des quinolines) pour y coupler la chaîne biotinylée. Le ligand final PDB-biotin a ensuite été obtenu après méthylation des quinolines. De mon côté, j’ai couplé la chaîne biotinylée sur un dérivé bromé de ttpy, puis ai complexé du cuivre(II) pour obtenir Cu-ttpy-biotin.
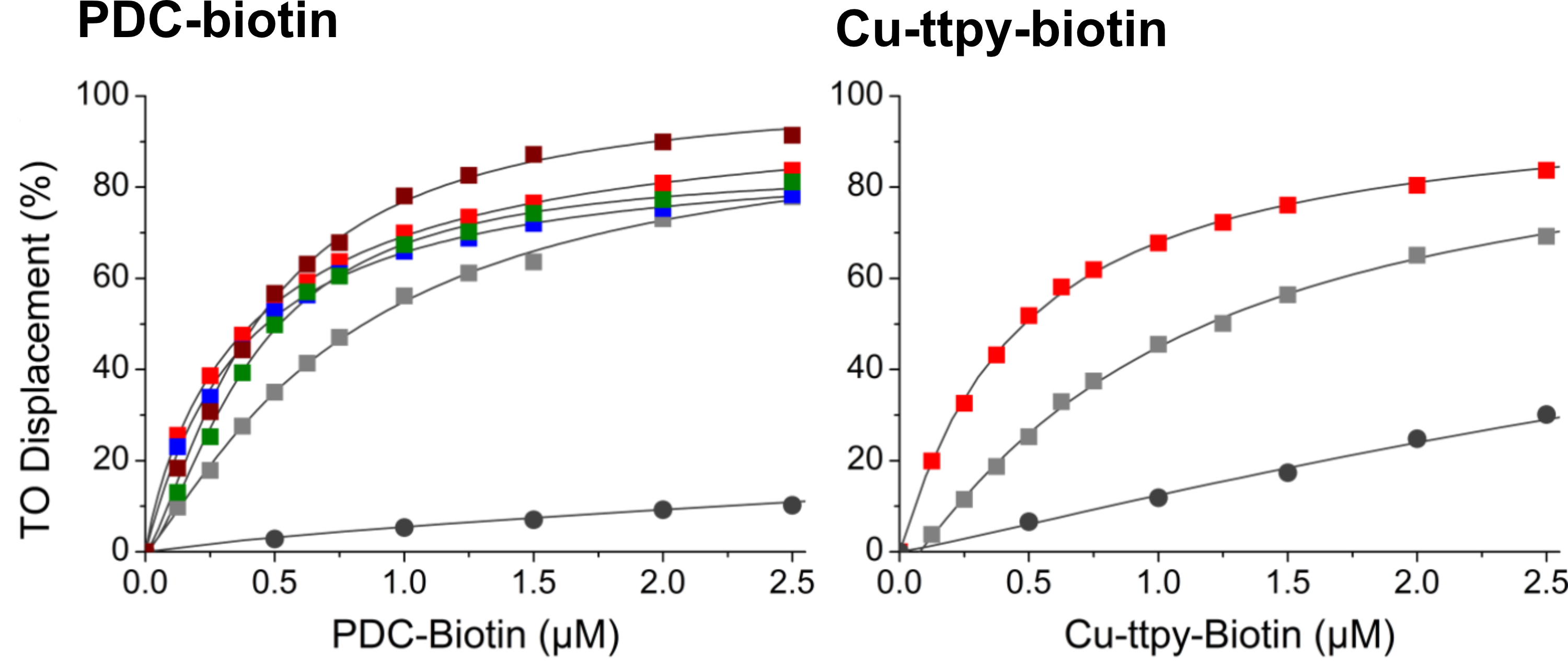
Les expériences HT-G4-FID ont confirmé que le ligand PDC‑biotin conserve une affinité élevée pour les G4s, comparé à 360A, ainsi qu’une sélectivité remarquable par rapport à l’ADN duplex (Figure 1.29). À l’inverse, Cu-ttpy-biotin a une affinité nettement plus faible pour les G4s et, en outre, est moins sélectif que Cu‑ttpy, ce qui en fait un moins bon candidat pour les expériences de SELEX.
Finalement, peu des aptamères sélectionnés (5 sur 80) étaient des G4s, ce qui a montré que des sites d’interactions inattendus peuvent être observés lors d’analyse de génomes ou transcriptomes entiers. La plupart des méthodes de caractérisation de l’affinité et sélectivité de ligands de G4 incluent peu de contrôles ; souvent seulement des doubles hélices. La sélectivité réelle des ligands de G4 est donc sans doute plus faible qu’on ne le pense, ce que démontre bien cette étude in vitro. Par extension, on peut penser que cela est pire dans les milieux in vivo.
1.4.2 Sélectivité de sondes fluorescentes : le test G4-POSCA
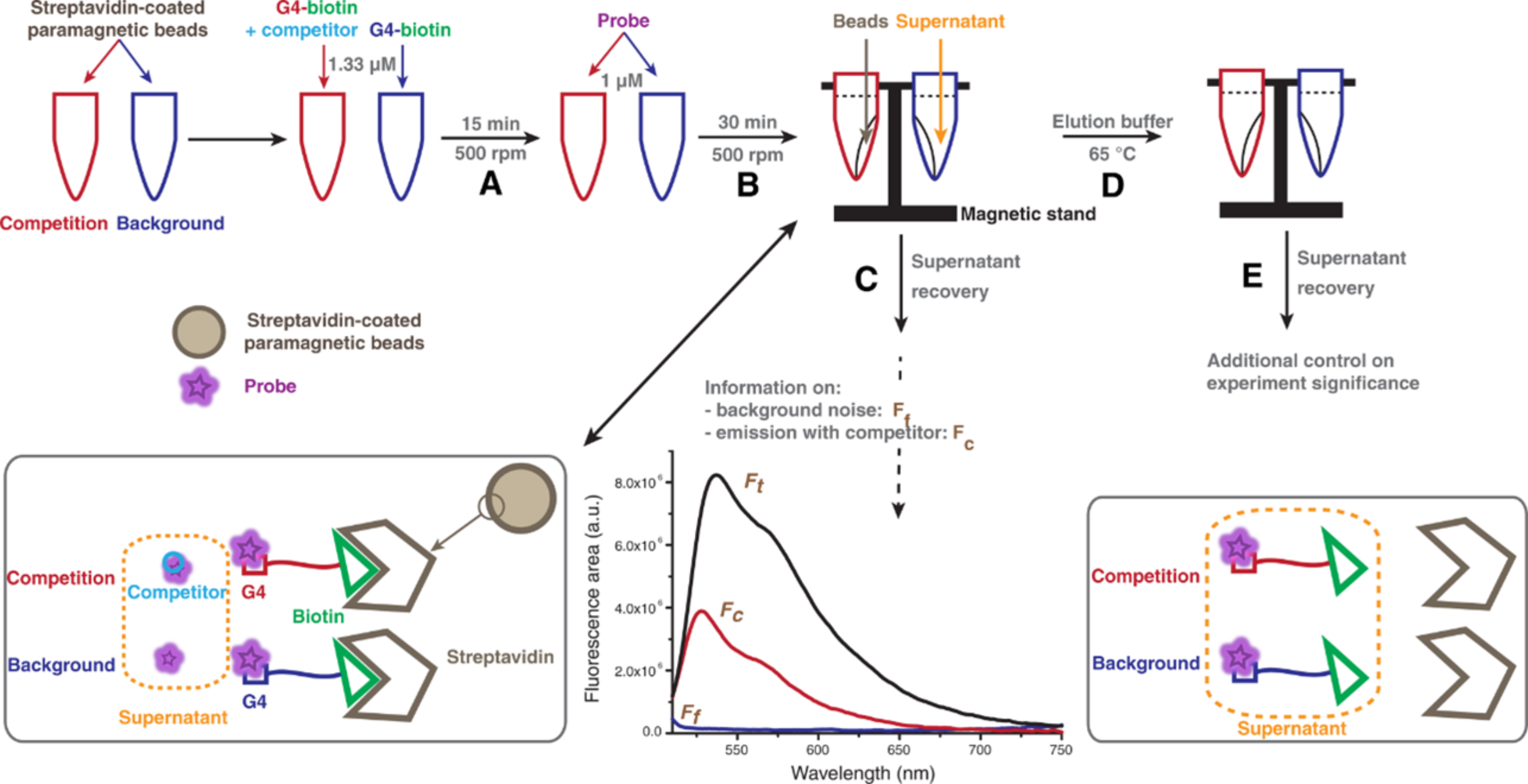
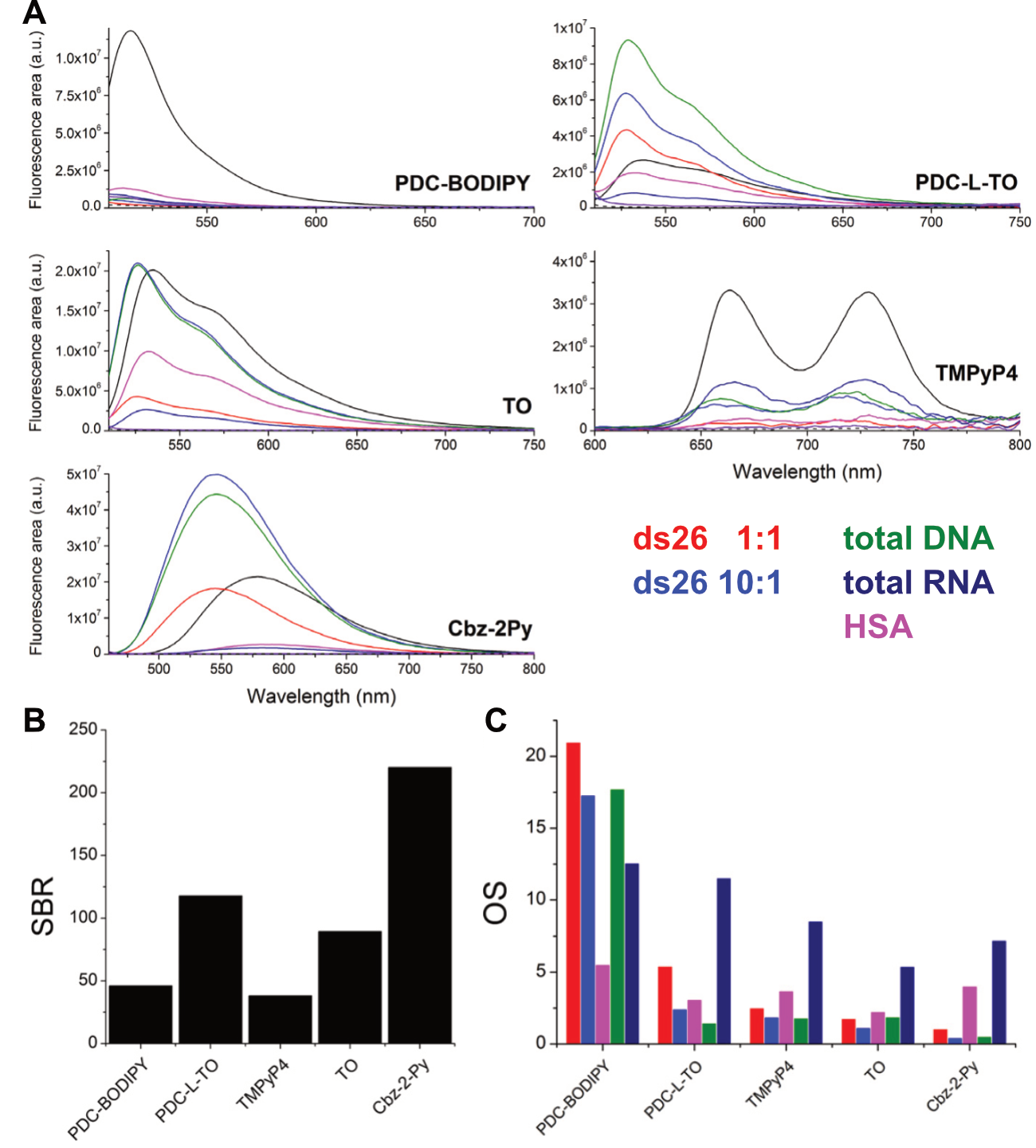
Comme nous l’avons vu ci-avant, je me suis beaucoup intéressé aux ligands fluorescents de G4 [84]. Cela m’a amené à concevoir une méthode originale, G4-POSCA, qui ne s’intéresse pas à proprement parlé à la sélectivité de petites molécules pour les G4s, mais de leur sélectivité optique (OS), c’est-à-dire de leur capacité à révéler la présence de G4s par fluorescence en présence d’autres cibles ‘compétitrices’ [78]. Un des grands avantages de cette méthode est d’ailleurs de pouvoir utiliser n’importe quel compétiteur, sans modification nécessaire, ici : différents excès de duplex, d’ADN total, d’ARN total, de protéine (la HSA) et même un autre G4.
La cible G4 biotinylée est d’abord mélangée ou non à ces compétiteurs (Figure 1.30A). Après incubation en présence du ligand fluorescent (Figure 1.30B), le G4 est immobilisé sur des billes magnétiques couvertes de streptavidine et le surnageant est récupéré (Figure 1.30C). On compare alors l’intensité de fluorescence du ligand en présence de G4 seul, avec celle venant d’une interaction avec le compétiteur (mesure de la sélectivité optique : surnageant du tube bleu dans la Figure 14) et lorsque le ligand est non lié (mesure du rapport signal-sur-bruit : surnageant du tube rouge). J’ai encadré une étudiante d’IUT, Margaux Legrand, lors de cette étude.
Pour le développement de cette méthode, j’ai utilisé plusieurs molécules fluorescentes avec des affinités, sélectivités et fluorescences diverses, que le test G4-POSCA a pu confirmer (Figure 1.31) :
PDC-BODIPY et PDC-L-TO sont deux dérivés de 360A, qui possède une grande affinité et une grande sélectivité pour les G4 [57, 89, 115]. Nous avons vu ci-dessus que a fonctionnalisation de 360A sur sa pyridine ne perturbe pas de manière significative son affinité pour les G4s (Section 1.4.1.3.4), ce qui est confirmé par l’excellente OS de PDC-BODIPY. En revanche, le TO peut se lier avec les G4s et les duplexes - ce qui diminue sensiblement la sélectivité de PDC-L-TO, mais ne fluoresce que lorsqu’il est lié ce qui lui donne un meilleur rapport signal-sur-bruit de fond (SBR).
Le dérivé de carbazole Cbz-2Py est une sonde fluorescente à deux photons, qui se lie préférentiellement au petit sillon de l’ADN duplex riche en AT, mais qui peut également se lier à des structures quadruplex avec un Kd de l’ordre du micromolaire. Cette faible sélectivité est visible ici (faible OS), tout comme les excellentes propriétés de fluorescence de cette molécule (meilleur SBR).
Le TMPyP4, une molécule très utilisée comme ligand de G4, qui a certes une bonne affinité pour ces derniers mais une sélectivité faible (voire inexistante) entre G4 et duplex. Cela est évident par FRET-melting, HT-G4-FID et dialyse compétititve [89, 116, 117], et se confirme ici.5
Le TO, qui n’est pas sélectif mais possède un bon SBR.
5 Si le lecteur doit retirer une seule information de ce manuscrit, c’est que TMPyP4 n’est pas un bon ligand de G4.
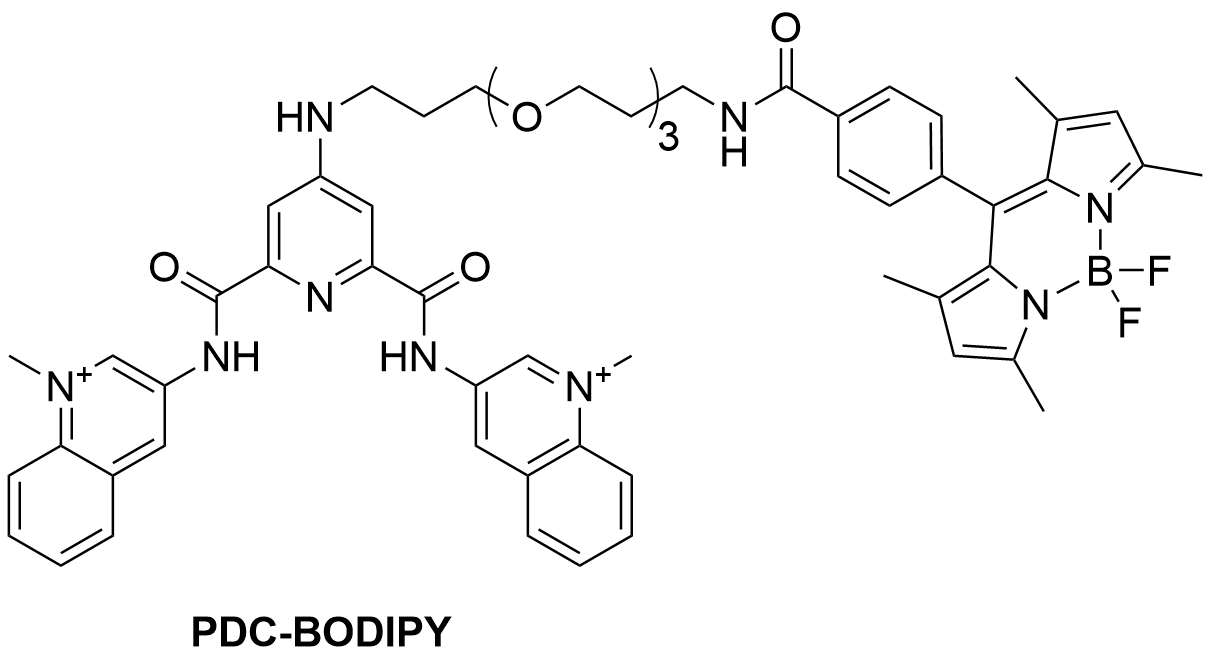
Si les deux derniers ligands sont commerciaux, les trois premiers ont été préparés au laboratoire. PDC-L-TO a été synthétisé avant mon arrivé par Peng Yang [118], Cbz-2Py par Blaise Dumat [119], un collègue doctorant (maintenant CR CNRS dans l’UMR7203 de l’ENS Paris), et enfin PDC-BODIPY par Reuben Ovadia (Master, Université de Montpellier ; désormais ingénieur brevets chez Servier) co-encadré avec Florian Hamon (Figure 1.32). À ma connaissance, la synthèse de ce dernier n’a jamais été publiée, à l’inverse de PhenDC3-bodipy [120].
1.5 Conclusion
Ce travail de thèse a combiné synthèse de ligands de G4 par chimie organométallique et organique, au développement d’outils d’analyse permettant de caractériser aux mieux ces ligands et leurs complexes, mais aussi d’en découvrir de nouveaux. Nous avons démontré que nos complexes de Pd2+ et Pt2+ interagissent avec les structures G4 par des mécanismes covalents et non covalents, et que l’oligohétéroaryle acyclique TOxaPy présente une sélectivité topologique inédite en ciblant préférentiellement les sillons plutôt que les tétrades. Parallèlement, la mise au point du test HT-G4-FID a permis le criblage et la caractérisation rapides d’un grand nombre de ligands potentiels. Ce chapitre illustre ainsi comment la conception rationnelle de nouvelles molécules, associée à des méthodes analytiques robustes, peuvent élargir le spectre des interactions ligand·G4. Comme un clin d’oeil du destin, ce qui sera sans doute mon dernier article publié avant de soutenir cette HDR revisitera ce thème, avec un brin de chance en prime.
D’un point de vue personnel, ces années passées à l’Institut Curie ont constitué une période particulièrement enrichissante, tant sur le plan scientifique qu’humain, et ont posé les fondations de la suite de mon parcours. J’y ai trouvé ma voie, pas en chimie organique, comme je l’imaginais au départ, mais avec une motivation renouvelée pour l’exploration des interfaces entre la chimie et les systèmes biologiques.


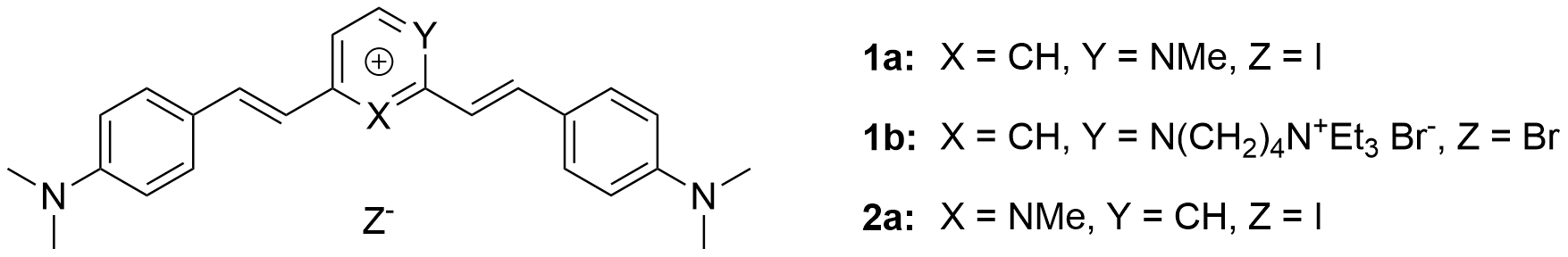
![Frontispiece du volume 19, numéro 4 de Chemistry – A European Journal [80]](resources/mcontent.jpg)